DNA 序列的分类模型
19 组 :王苗苗,吴冲,赵林娜
摘要
本文针对 DNA 序列的分类问题,建立了基于碱基序列特征参数的分类模型。
针对问题一:通过对已知 20 个人工 DNA 序列分析,分别从碱基的含量、碱基串的含
量、碱基的排列顺序三个方面,提取出特征指标,建立了基于 Fisher 的分类方法和 RBF
神经网络的分类方法的两个分类模型。基于 DNA 序列的特征,从碱基含量出发,提取了
g,t 的百分比的特征指标;从碱基串的含量出发,考虑 DNA 序列是由众多三联体组成,
提取了 DNA 无极性,极性带正电,极性带负电氨基酸的百分比的特征指标;从碱基的排
列顺序出发,基于 DNA 序列的空间表示,提取了碱基序列在 和 平面上拟合直线
的斜率和点集均值的特征指标,并将这一系列指标有机组合为一综合指标,在此基础上,
提出了基于综合指标的 fisher 分类模型和 RBF 分类模型,并从复杂度,鲁棒性,适用
性等方面评价了模型的优劣。
针对问题二:建立了基于多指标的高维超椭球的距离比和基于碱基序列排列的 Z 曲
线空间距离比两个评价指标的评价模型,利用评价模型对分类结果进行分析。从综合指
标的角度,将两类 DNA 序列在 9 维空间上的类间距离与类内平均距离之比做为指标一;
将两类 DNA 序列排列的空间 Z 曲线的类间距离与类内平均距离之比做为指标二。在此基
础上,对 fisher 分类法和 RBF 分类法进行了优劣评价,并验证了模型的合理性。最后选
择利用 RBF 网络对对 21-40 组待测 DNA 序列进行分类,得到如下结果:
类别
A 类
B 类
DNA 序列的序号
23,25,27,29,34,35,36,37,39
21,22,24,26,28,30,31,32,33,38,40
在此基础上,分析了 RBF 分类法对 spread 参数的灵敏度,fisher 对阀值权重的灵敏度。
针对问题三:采用问题二较为满意的分类模型对 185 个自然 DNA 序列分成 A、B 两类,
利用评价模型对 185 个自然 DNA 序列的分类结果分析,发现两个评价的指标很小,由于
基于 RBF 的分类模型对 185 个 DNA 序列分类的结果不满意,考虑到 185 个自然 DNA 序列
分类情况不是很明显,提出了聚类分析模型,从评价指标分析聚类分析的均比基于 RBF
的分类模型分类结果好。得出 185 个自然 DNA 序列的分类结果。
关键字:DNA 序列 特征提取 评价指标 Fisher 分类 RBF 神经网络 聚类分析
xoszos�
本文针对 DNA 序列分类问题建立了相应的数学模型。
针对问题一:基于 DNA 序列的特征,从碱基含量出发,提取了 g,t 的百分比的特征指标;
从碱基串的含量出发,考虑 DNA 序列是由众多三联体组成,提取了 DNA 无极性,极性带
正电,极性带负电氨基酸的百分比的特征指标;从碱基的排列顺序出发,基于 DNA 序列
的空间表示,提取了投影平面的斜率与均值的特征指标。并将这一系列指标有机组合为
一综合指标,在此基础上,提出了基于综合指标的 fisher 分类法和基于综合指标 RBF
分类法,并从复杂度,鲁棒性,适用性等方面评价了模型。
针对问题二:提出了基于多指标的高维超椭球的离散平均距离比评价指标和基于碱基序
列排列的 Z 曲线空间距离比评价指标。在此基础上,对 fisher 分类法和 RBF 分类法进行
了优劣评价,并验证了模型的合理性。最后选择利用 RBF 网络对对 21-40 组待测 DNA 序
列进行分类,得到如下结果:
类别
A 类
B 类
DNA 序列的序号
23,25,27,29,34,35,36,37,39
21,22,24,26,28,30,31,32,33,38,40
在此基础上,分析了 RBF 分类法对 spread 参数的灵敏度,fisher 对阀值权重的
针对问题三:分析了 DNA 序列数据,提出了聚类分析模型,利用此模型对 185 组 DNA 序
列进行分类。利用评价指标,对比了 RBF 分类法,fisher 分类法与聚类分类法的优劣。
发现对于数据量较大,没有初始训练样本的 DNA 序列,聚类分析方法能得到较优的分类
结果。
关键字:DNA 序列 特征提取 评价指标 Fisher 分类 RBF 神经网络 聚类分析
�
一、 问题重述
21 世纪既是生命科学的时代,也是信息的时代。随着基因研究和信息技术的发展,
有关核酸碱基序列的数据呈指数增长,这些序列上不仅包含着制造人类所有蛋白质的信
息, 还包含着将这些蛋白质装配成生物体的调控信息。因此,DNA 序列的研究对人类社
会遗传进化的研究和生物学的发展有着重大的意义。
DNA 虽然仅由腺嘌呤、鸟嘌呤、胞嘧啶、胸腺嘧啶四种碱基按一定顺序排列而成,
但是却含有这世界上最巨量的信息,是一部人类至今还无法读懂 “天书”,但是人类也
发现了 DNA 序列中的一些规律和结构特征。例如,在全序列中有一些是用于编码蛋白
质的序列片段,即由这 4 个字符组成的 64 种不同的 3 字符串,其中大多数用于编码构
成蛋白质的 20 种氨基酸。又例如,在不用于编码蛋白质的序列片段中,A 和 T 的含量
特别多些。此外,利用统计的方法还发现序列的某些片段之间具有相关性。这些发现让
人们相信,DNA 序列中存在着局部的和全局性的结构,充分发掘序列的结构对理解 DNA
全序列是十分有重要的。针对题目要求建立恰当的数学模型,解决以下问题:
问题一:有已知类别的 20 条碱基序列,其中序列标号 1—10 为 A 类,11-20 为 B
类。请从中提取特征,构造至少 2 种分类方法,并评价分类方法的优劣。
问题二:利用比较精确地分类方法,对附件 1 中另外 20 个未标明类别的人工序列(标
号 21—40)进行分类。
问题三:用上面的分类方法对附件 2 中 185 个自然 DNA 序列进行分类。
二、 问题分析
伴随信息时代的快速发展和基因技术的不断深入,DNA 碱基序列的研究成为当今时
代的热门话题。DNA 虽有四种碱基组成,但却含有巨量的生命遗传信息,对人类社会的
健康发展起着十分关键的作用。本文针对碱基序列分类问题,建立 Fisher、RBF 和系统
聚类三种模型,对 205 个未知类别的 DNA 序列进行合理的分类。
针对问题一,结合生物学知识,根据 A、B 两类的碱基序列,从中提取碱基含量、
碱基串含量、碱基的排列顺序三个因子作为特征指标,通过分析比较 A、B 两类碱基序
列的特点,建立 Fisher 和 RBF 模型作为分类方法,并对两种模型的优缺点分进行合理的
分析。
针对问题二:对未知类别的人工序列,利用问题一建立的 Fisher 和 RBF 模型,得到
两种分类结果,并对 RBF 与 BP 模比较、Fisher 模型和 RBF 模型的灵敏度进行分析,为
了获取较好的序列分类,提出基于多指标的高维超椭球的距离比和多指标的高维超椭球
的距离比两种指标,
针对问题三:对未知类别的自然序列,由于自然 DNA 序列含有编码区和非编码区,
所以不能直接运用问题一建立的 Fisher 和 RBF 模型,为对自然序列进行合理分类,建立
�
系统聚类模型,并对序列进行合理分类。利用问题二为中的评价指标,对 fisher、RBF
与聚类分类三种模型进行优劣分析。
三、模型假设
(1)、假设题中所给的碱基序列均属于 A 类或 B 类中的一种。
(2)、假设题中所给的 DNA 片段在截取时起始片段至多相差两个碱基。
(3)、假设 DNA 在合成氨基酸的过程中,基因正常表达,没有基因突变和其他因素的干
扰。
(4)、假设 DNA 在合成氨基酸的过程中,碱基剩余个数小于 3 时舍去不考虑。
四、符号说明
碱基 A 的百分含量
碱基 G 的百分含量
碱基 C 的百分含量
碱基 T 的百分含量
某类样品第 个指标值
第
类序列的对应点群的均值
DNA 序列指标类内的离散程度
两个类 DNA 序列一维投影的均值
一维投影的类间离散程度
一维投影的类内离散程度
输出层第 个神经元的输出值
第 、 个神经元间的权值
离散距离比
类内曲线到中心曲线的空间平均距离
空间距离比
样品 , 之间的距离
时刻类中第 i 个样品
\
\
\
\
\
\
\
\
\
\
\
\
\
\
\
\
\
papgpcpcixiim(1,2)iisimABSiSkyjkjwkjFJidZJ(,)ijdxxixjxtiX()t�
类 中样品的离差平方和
与 之间的离差平方和距离
\
\
五、模型的建立与说明
5.1、问题一的模型建立与求解
5.1.1、DNA 序列的特征提取
根据题意, 本文首先要提取出A类和B类的DNA序列特征, 给出每类特征的数学表
示, 最后选择出合适精确的分类方法以对未知序列进行分类。
对于20条已知类别的DNA 序列,结合生物学背景和建模需要,一方面提取的特征
要具有生物学意义,另一方面所提取特征能运用于数学模型,因此, 本文选择以下三
个因子作为反映两类序列特征的指标:
碱基的含量,反映了该序列的基本组成;
碱基串的含量,反映了该序列的基本形式;
碱基的排列顺序,反映了该序列的基本结构。
1)、特征一:碱基含量
DNA 序列由 A、G、C、T 四种碱基组成,碱基含量在生物学中有重要的意义,因为 DNA
在编码蛋白质的区段 C 和 G 的含量较高,在非编码蛋白质区段 A 和 T 的含量较高,因此
可通过判断某些碱基的含量对 DNA 序列进行分类。
设碱基序列中 A、G、C、T 的百分含量分别为
,由此可得到一组四
,所以可用三维向
维向量,但是
量
代替四维向量,[1]这组三维向量有着重要的作用,由上面所述的生物学
意义,可根据碱基含量的组成对未知的 DNA 序列的结构进行深入的研究。已知 A 类和 B
类的碱基含量如下表:
呈线性相关,即
1
2
3
4
5
11
12
表格 1 四种碱基的百分含量
A
0.2973
0.27027
0.27027
0.42342
0.23423
0.35455
0.32727
c
0.17117
0.16216
0.21622
0.10811
0.23423
0.045455
0.027273
g
0.3964
0.41441
0.45045
0.18018
0.42342
0.1
0.14545
t
0.13514
0.15315
0.063063
0.28829
0.10811
0.5
0.5
tstGpqDPGqGpapgpcpt、、、papgpcpt、、、1papgpcpt(,,)papgpc�
13
14
15
0.25455
0.1
0.3
0.081818
0.29091
0
0.12727
0.11818
0.063636
0.51818
0.5
0.64545
(说明:该表中只列出了 A 类的前五条序列和 B 类的前五条序列)
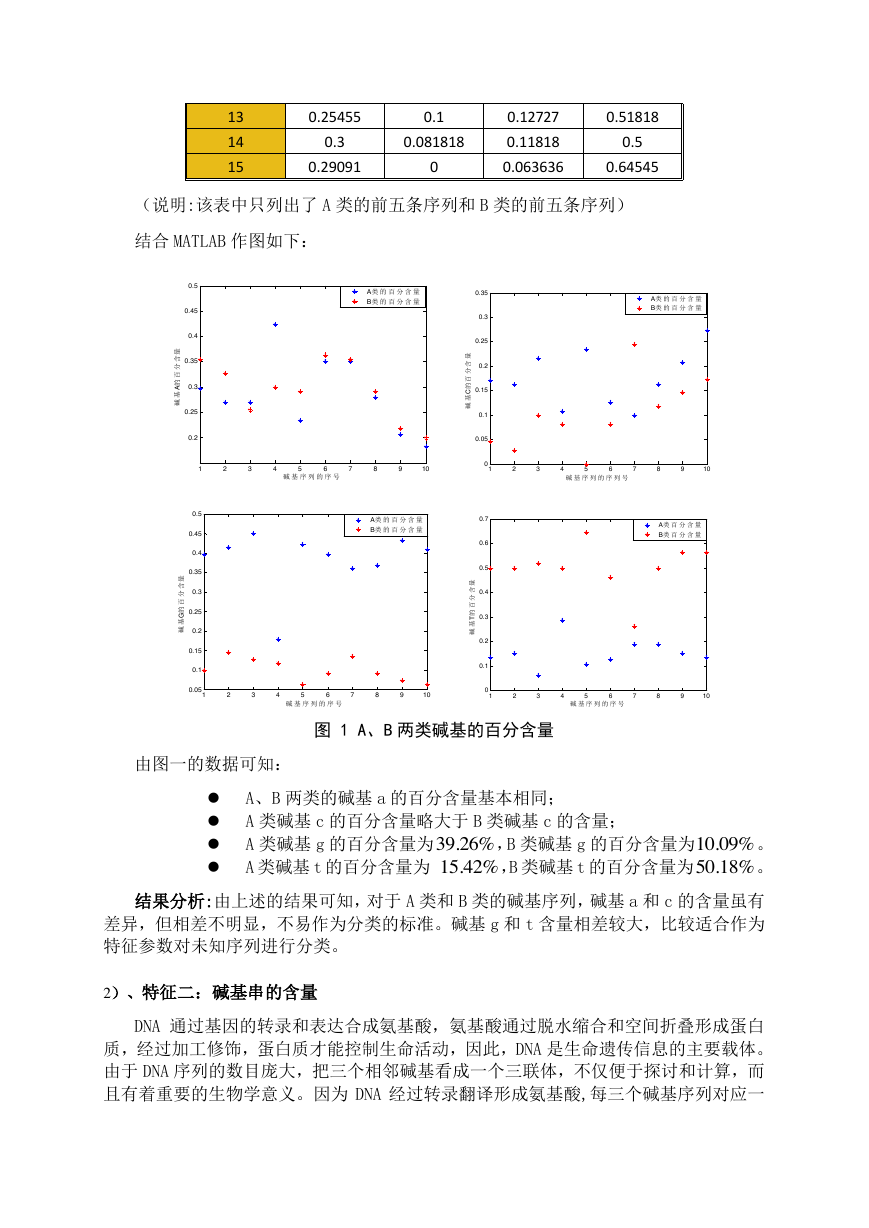
结合 MATLAB 作图如下:
图 1 A、B 两类碱基的百分含量
由图一的数据可知:
A、B 两类的碱基 a 的百分含量基本相同;
A 类碱基 c 的百分含量略大于 B 类碱基 c 的含量;
A 类碱基 g 的百分含量为
A 类碱基 t 的百分含量为
,B 类碱基 g 的百分含量为
,B 类碱基 t 的百分含量为
。
。
结果分析:由上述的结果可知,对于 A 类和 B 类的碱基序列,碱基 a 和 c 的含量虽有
差异,但相差不明显,不易作为分类的标准。碱基 g 和 t 含量相差较大,比较适合作为
特征参数对未知序列进行分类。
2)、特征二:碱基串的含量
DNA 通过基因的转录和表达合成氨基酸,氨基酸通过脱水缩合和空间折叠形成蛋白
质,经过加工修饰,蛋白质才能控制生命活动,因此,DNA 是生命遗传信息的主要载体。
由于 DNA 序列的数目庞大,把三个相邻碱基看成一个三联体,不仅便于探讨和计算,而
且有着重要的生物学意义。因为 DNA 经过转录翻译形成氨基酸,每三个碱基序列对应一
123456789100.20.250.30.350.40.450.5碱基序列的序号碱基A的百分含量A类的百分含量B类的百分含量1234567891000.050.10.150.20.250.30.35碱基序列的序列号碱基C的百分含量A类的百分含量B类的百分含量123456789100.050.10.150.20.250.30.350.40.450.5碱基序列的序号碱基G的百分含量A类的百分含量B类的百分含量1234567891000.10.20.30.40.50.60.7碱基序列的序号碱基T的百分含量A类百分含量B类百分含量39.26%10.09%15.42%50.18%�
个氨基酸,所以可通过研究 DNA 序列翻译的各种氨基酸的百分含量来判断序列的类别,
但是因为基本氨基酸有 20 种,显然作为特征参数进行探讨过于复杂,本文为便于计算,
将 20 种氨基酸根据极性和带电性分成无极性、极性不带电、极性带正电、极性带负电
四种氨基酸。
四种氨基酸求解:
Step1:考虑到题中所给片段是截取的部分片段,根据假设截取时起始片段至多相差
两个碱基,在计算时本文针对起始片段分别相差 0 个、1 个、2 个碱基三种情况分别移
位计算三联体的数量。
Step2:三种情况取平均值得到最后的各种三联体数量,再除以该序列碱基总数,得
到各种三联体的百分含量。
Step3:根据生物学知识,三个碱基组合可形成 64 种密码子,其中 61 种翻译形成氨
基酸,剩余三种为终止密码子,根据氨基酸的极性和带电性,可得到四种氨基酸分别对
应的 DNA 碱基序列,结果见附录。
四种氨基酸含量的结果:
通过编程求解得到每条碱基序列中无极性、极性不带电、极性带正电、极性带负电
四种氨基酸的百分含量如下表:
表格 2 氨基酸的含量
无极性 极性不带电 极性带正电 极性带负电
0.581761
0.2955975
0.0849057
0.0377358
0.6057692
0.2596154
0.1153846
0.0192308
0.6603774
0.2264151
0.1037736
0.009434
0.5242718
0.3398058
0.1165049
0.0194175
0.6
0.2190476
0.1333333
0.047619
0.647619
0.2095238
0.1428571
0
0.6078431
0.2843137
0.0882353
0.0196078
0.5428571
0.2952381
0.1238095
0.0380952
0.5142857
0.2666667
0.1904762
0.0285714
0.5111111
0.2412698
0.2095238
0.0380952
0.4631579
0.2947368
0.2315789
0.0105263
0.4315789
0.3508772
0.2070175
0.0105263
0.3573883
0.3402062
0.2714777
0.0309278
0.3505155
0.3298969
0.2680412
0.0515464
0.3231293
0.2142857
0.462585
0
0.4623656
0.2795699
0.1935484
0.0645161
0.4915825
0.3030303
0.1245791
0.0808081
0.4042553
0.3297872
0.2234043
0.0425532
0.2947368
0.2842105
0.3263158
0.0947368
0.2385965
0.2210526
0.3824561
0.1578947
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
�
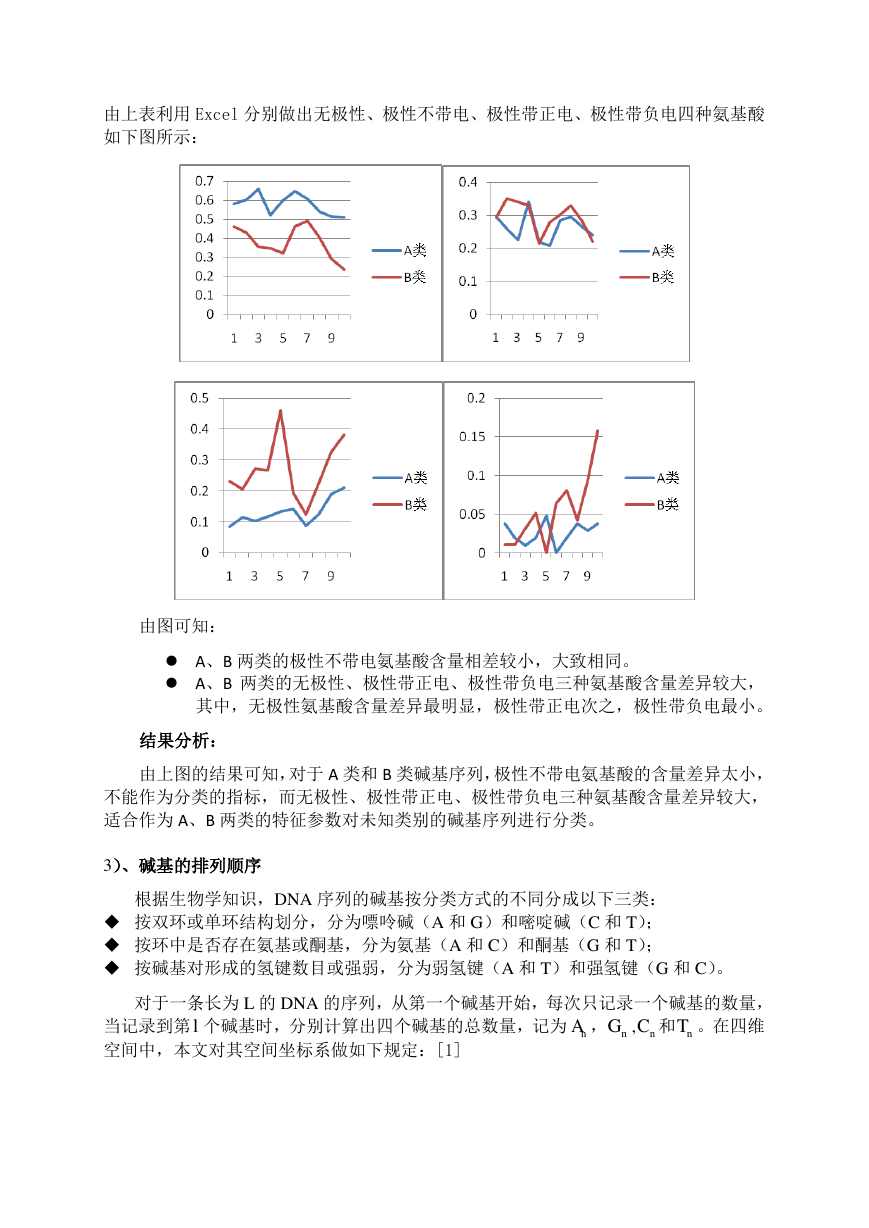
由上表利用 Excel 分别做出无极性、极性不带电、极性带正电、极性带负电四种氨基酸
如下图所示:
由图可知:
A、B 两类的极性不带电氨基酸含量相差较小,大致相同。
A、B 两类的无极性、极性带正电、极性带负电三种氨基酸含量差异较大,
其中,无极性氨基酸含量差异最明显,极性带正电次之,极性带负电最小。
结果分析:
由上图的结果可知,对于 A 类和 B 类碱基序列,极性不带电氨基酸的含量差异太小,
不能作为分类的指标,而无极性、极性带正电、极性带负电三种氨基酸含量差异较大,
适合作为 A、B 两类的特征参数对未知类别的碱基序列进行分类。
3)、碱基的排列顺序
根据生物学知识,DNA 序列的碱基按分类方式的不同分成以下三类:
按双环或单环结构划分,分为嘌呤碱(A 和 G)和嘧啶碱(C 和 T);
按环中是否存在氨基或酮基,分为氨基(A 和 C)和酮基(G 和 T);
按碱基对形成的氢键数目或强弱,分为弱氢键(A 和 T)和强氢键(G 和 C)。
对于一条长为 L 的 DNA 的序列,从第一个碱基开始,每次只记录一个碱基的数量,
当记录到第 个碱基时,分别计算出四个碱基的总数量,记为 , , 和 。在四维
空间中,本文对其空间坐标系做如下规定:[1]
lnAnGnCnT�













 2023年江西萍乡中考道德与法治真题及答案.doc
2023年江西萍乡中考道德与法治真题及答案.doc 2012年重庆南川中考生物真题及答案.doc
2012年重庆南川中考生物真题及答案.doc 2013年江西师范大学地理学综合及文艺理论基础考研真题.doc
2013年江西师范大学地理学综合及文艺理论基础考研真题.doc 2020年四川甘孜小升初语文真题及答案I卷.doc
2020年四川甘孜小升初语文真题及答案I卷.doc 2020年注册岩土工程师专业基础考试真题及答案.doc
2020年注册岩土工程师专业基础考试真题及答案.doc 2023-2024学年福建省厦门市九年级上学期数学月考试题及答案.doc
2023-2024学年福建省厦门市九年级上学期数学月考试题及答案.doc 2021-2022学年辽宁省沈阳市大东区九年级上学期语文期末试题及答案.doc
2021-2022学年辽宁省沈阳市大东区九年级上学期语文期末试题及答案.doc 2022-2023学年北京东城区初三第一学期物理期末试卷及答案.doc
2022-2023学年北京东城区初三第一学期物理期末试卷及答案.doc 2018上半年江西教师资格初中地理学科知识与教学能力真题及答案.doc
2018上半年江西教师资格初中地理学科知识与教学能力真题及答案.doc 2012年河北国家公务员申论考试真题及答案-省级.doc
2012年河北国家公务员申论考试真题及答案-省级.doc 2020-2021学年江苏省扬州市江都区邵樊片九年级上学期数学第一次质量检测试题及答案.doc
2020-2021学年江苏省扬州市江都区邵樊片九年级上学期数学第一次质量检测试题及答案.doc 2022下半年黑龙江教师资格证中学综合素质真题及答案.doc
2022下半年黑龙江教师资格证中学综合素质真题及答案.doc