细胞凋亡蛋白 Bcl-2 的分子动力学模拟
学号:1030402016
姓名:徐优俊
一、背景:
在细胞凋亡的相关调控基因的研究方面,Bcl-2 基因是目前研究的最深入、最广泛的
凋亡调控基因之一。最近,在肝细胞中发现了乙型肝炎病毒(Hepatitis B virus, HBV)的两
种主要靶标(Bcl-2 和 Bcl-xL);HBV 编码一种致病的促进肿瘤产生的蛋白 HBx,HBx 的
宿主靶标是两种小分子蛋白 Bcl-2 和 Bcl-xL,HBx 使用一种特殊的基序---它是一小段氨
基酸序列,类似于在一些导致细胞死亡的蛋白中发现的序列---来与靶标 Bcl-2 和 Bcl-xL
相互作用,促进宿主细胞中钙离子水平提高。钙离子水平提高随后触发病毒 HBV 复制
和细胞死亡。这种基序发生基因突变时,HBx 结合到 Bcl-2 和 Bcl-xL 蛋白上,并阻止病
毒复制。类似地,当人肝细胞中 Bcl-2 或 Bcl-xL 蛋白被敲降或功能减弱时,HBx 更不能
够导致被感染的细胞内的钙离子水平增加和病毒复制。
二、实验步骤:
1) 从 PDB 网站(http://www.rcsb.org/pdb/home/home.do)上下载蛋白结构 4IEH.pdb 文
件。
4IEH.pdb(Bcl-2)的结构:
Fig1: Licorice,Tube and NewCartoon representations of Bcl-2 protein
相关信息介绍:
Bcl-2 基因家族是目前广泛研究的一类细胞凋亡相关基因,其表达和调控是影响细胞
凋亡的关键因素之一,在细胞凋亡信号转导途径中发挥质量要作用。通过上调促凋亡的
Bcl-2 的基因表达,从而在转录水平调节凋亡相关蛋白的表达以影响凋亡的发生。Bcl-2
家族成员都含有 1-4 个 Bcl-2 同源结构域(BH1-4),并且通常有一个羧端跨膜结构域。
Bcl-2 的网络结构图:
�
Fig2: This is the evidence view. Different line colors represent the types of evidence for the association.
including
B-cell CLL/lymphoma 2; Suppresses apoptosis
factor-dependent lymphohematopoietic and neural cells. Regulates cell death by controlling the
mitochondrial membrane permeability. Appears to function in a feedback loop system with
caspases. Inhibits caspase activity either by preventing the release of cytochrome c from the
mitochondria and/or by binding to the apoptosis-activating factor.
in a variety of cell
systems
2) 用 UltraEdit 打开 4IEH.pdb 文件,将 HETATM 一段(除去其中的水),从上图看出,
该蛋白结构存在两条链,除去较短的链,保存为 4IEH_2.pdb 文件。
3) GROMACS 实验准备阶段:
1) Pdb2gmx,建立该蛋白的拓扑文件,其中选择 10(43a2 力场)、2(SPC/E
水),生成 4IEH_2_1.pdb 和 4IEH_2_1.top 文件。
>pdb2gmx -ignh -f 4IEH_2.pdb -o 4IEH_2_1.pdb -p 4IEH_2_1.top
-f: 指定你的坐标文件,可以是 pdb、gro、tpr 等等包含有分子坐标的文件;
-o: 输出文件,也就是处理过的分子坐标文件,同样可以是 pdb、gro、g96
等文件类型;
拓扑;
-p:输出拓扑文件。pdb2gmx 读入力场文件,根据坐标文件建立分子系统的
-ignh:舍弃分子文件中的 H 原子,因为 H 原子命名规则多,有的力场不认;
2) Editconf, 为蛋白质加上 1.5nm 厚度的盒子
>editconf -bt cubic -f 4IEH_2_1.pdb -o 4IEH_2_2.gro -d 1.5
-bt:盒子类型,有正方型,长方形,八面型等等;
-f :指定你的坐标文件;
-o :输出文件,即放进盒子里面的分子系统。
-d :分子离盒子表面的最短距离(nm)。
3) Genbox, 向上述的盒子中添加水分子,选择 SPC 模式的水溶液,生成溶质+
�
溶液体系的 4IEH_2_3.pdb 和 4IEH_2_1.top 文件
>genbox -cp 4IEH_2_2.gro -cs spc216.gro -o 4IEH_2_3.pdb -p
-cp:带盒子参数的分子坐标文件,也就是 Editconf 的输出文件;
-cs:添加的水分子模型,如 spc216、spce、tip3p、tip4p 等;
-o: 输出坐标文件,就是添加水分子之后的分子坐标文件,默认是.gro 文件,
4IEH_2_1.top
但是也可以输出其他文件格式,如 pdb;
-p: 系统拓扑文件。
4) Make_ndx,生成一个仅包含蛋白质分子的索引文件;
>make_ndx -f 4IEH_2_3.pdb -o 4IEH_2_4_pro.ndx
-f:指定你的坐标文件;
-o:输出 protein 的索引文件
运行上述代码过程中选择 1(protein);
ca.itp
>genrestr -f 4IEH_2_3.pdb -n 4IEH_2_4_pro.ndx -o ca.itp
-f:指定你的坐标文件;
-n:输入上述得出的 protein 的索引文件;
-o:输出仅包含某种原子的文件
运行上述代码过程中选择 3(C-alpha)的选项;
5) Genrestr, 在 protein 索引文件基础上生成一个仅包含 Ca 原子的索引文件
3) GROMACS 实验核心阶段:
主要步骤:在进行能量最优化之前,先用 GROMACS 的预处理程序 grompp 处理所有
输入文件。grompp 预处理拓扑文件(.top),坐标文件(.gro)和一个参数
文件(.mdp),然后输出一个二进制拓扑文件(.tpr)。再对前面的结果进行
动力学模拟,运行 mdrun 命令;
命令的参数介绍: grompp
mdrun
-f:所要输入的参数文件;
-c:所要输入的坐标文件(*.pdb);
-p:输入对应的拓扑文件;
-o:生成二进制拓扑文件,是运行 mdrun 命令的输入文件;
-s:输入预处理的 tpr 文件;
-o:输出新的拓扑文件;
-e:输出能量文件;
-g:运行该命令时的一些步骤信息;
-v:在终端显示运行的进度信息;
1) 对该蛋白的结构进行分步能量最小化。
A 固定 C-alpha 原子(mdp 文件中修改 define=-DCA),采用 steep 算法(修改
integrator=steep) , 进 行 1000 步 能 量 最 小 化 (nstep=1000), 加 入 参 数
coulombtype = PME fourierspacing = 0.12,用 PME 来计算静电库仑
力;
>grompp -f em6A.mdp -c 4IEH_2_3.pdb -p 4IEH_2_1.top -o 4IEH_2_6A.tpr
预处理出来的结果:电负性为-4.999999,为了平衡电荷,使体系不崩塌,向
SOL 体系中加入 5 个 Na+中和:
>genion -s 4IEH_2_6A.tpr -o 4IEH_2_6_ion.pdb
-s:输入拓扑文件;
-o:输出加入离子后的坐标文件(*.pdb);
-pname: 输入加入阳离子的名称,阴离子为-nname;
-pname NA+ -np 5
�
-np:输入加入阳离子的个数,阴离子为-nn;
在运行上述代码过程中选择 13(SOL)体系;
加入离子后,拓扑结构文件(*.top)需要修改: 在拓扑文件最后的的
“molecules”一栏,添加“NA
5”,然后在 SOL 数量中减 5。重新保存;
重新进行预处理:
>grompp
4IEH_2_1.top -o 4IEH_2_6A.tpr
>mdrun
4IEH_2_6A.log -c 4IEH_2_6A_em.gro –v
-po mdout6A.mdp
4IEH_2_6_ion.pdb
4IEH_2_6A.edr
4IEH_2_6A.tpr
4IEH_2_6A.tpr
em6A.mdp
-o
-c
-p
-g
-f
-s
-e
由上图
可知,
用
steep
算法跑
了 1000
步后未
收敛,
所以再
次进行
steep
算法;
grompp
-f em6A.mdp -po mdout6A.mdp -c 4IEH_2_6A_em.gro -p 4IEH_2_1.top -o
4IEH_2_6A.tpr
mdrun
4IEH_2_6A.log -c 4IEH_2_6A_em.gro -v
再次经
过
steep
算 法
后,在
跑 了
135 步
后 收
敛;
4IEH_2_6A.edr
4IEH_2_6A.tpr
4IEH_2_6A.tpr
-o
-g
-e
-s
B 放
开
所
有
原
子(mdp 文件中修改 define=-DFLEX_SPC),采用 l-bfgs 算法(修改
integrator=l-bfgs),进行 3000 步的能量最小化(nsteps
=
3000),并修改 emtol=200,加入参数 coulombtype = PME fourierspacing
�
= 0.12,用 PME 来计算静电库仑力。
em6B.mdp -c 4IEH_2_6A_em.gro
>grompp -f
4IEH_2_6B.tpr
>mdrun
4IEH_2_6B.log -c 4IEH_2_6B_em.gro –v
4IEH_2_6B.tpr
-o
-s
-p 4IEH_2_1.top -o
-e
4IEH_2_6A.edr
-g
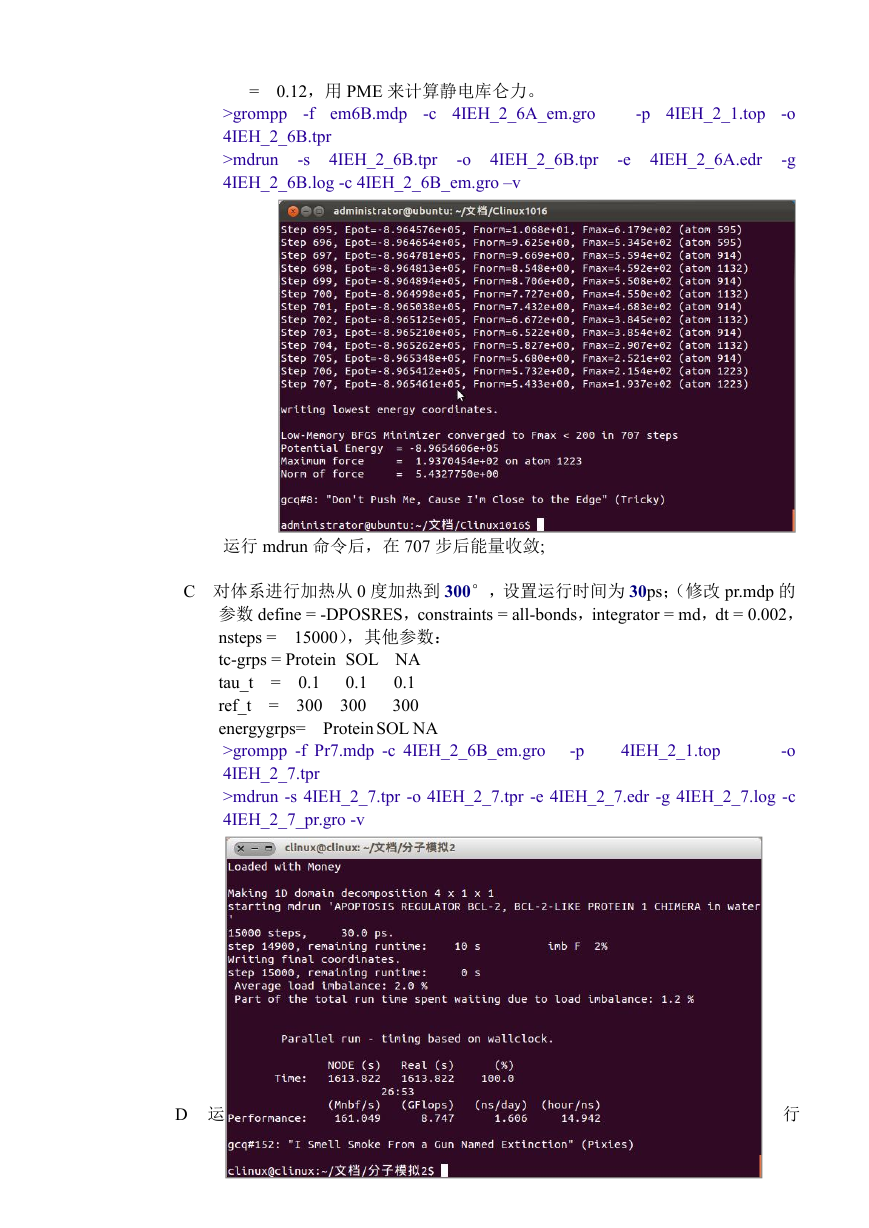
4IEH_2_6B.tpr
运行 mdrun 命令后,在 707 步后能量收敛;
C 对体系进行加热从 0 度加热到 300°,设置运行时间为 30ps;(修改 pr.mdp 的
参数 define = -DPOSRES,constraints = all-bonds,integrator = md,dt = 0.002,
nsteps = 15000),其他参数:
tc-grps = Protein SOL NA
0.1
tau_t = 0.1
ref_t = 300
300
energygrps= Protein SOL NA
>grompp -f Pr7.mdp -c 4IEH_2_6B_em.gro
4IEH_2_7.tpr
>mdrun -s 4IEH_2_7.tpr -o 4IEH_2_7.tpr -e 4IEH_2_7.edr -g 4IEH_2_7.log -c
4IEH_2_7_pr.gro -v
4IEH_2_1.top
0.1
300
-p
-o
D 运
行
�
100ps 的分子动力学模拟。(修改参数 nsteps = 50000,生成 md1.mdp)
>grompp -f md1.mdp -c 4IEH_2_7_pr.gro
4IEH_2_8.tpr
>mdrun -s 4IEH_2_8.tpr -o 4IEH_2_8.tpr -e 4IEH_2_8.edr -g 4IEH_2_8.log -c
4IEH_2_8_md.gro -v
-p 4IEH_2_1.top
-o
4) 对模拟得到的轨迹文件.trr 文件进行分析:
A 用 g_rms,计算蛋白质 Ca 原子随时间的 RMSD 结果,画图;
-f
-n 4IEH_2_4_pro.ndx -o
4IEH_2_8.tpr
4IEH_2_8.tpr.trr
>g_rms -s
4IEH_2_9_RMSD.xvg
> 4IEH_2_9_RMSD.xvg
-s:输入拓扑文件;
-f:输入运行 md 生成的轨迹文件;
-n:输入蛋白的索引文件;
-o:输出 RMSD 的结果文件;
运行上述代码过程中选择 1(protein)、3(C-alpha)的体系;
xmgrace:解析 xvg 文件,得出图像文件;
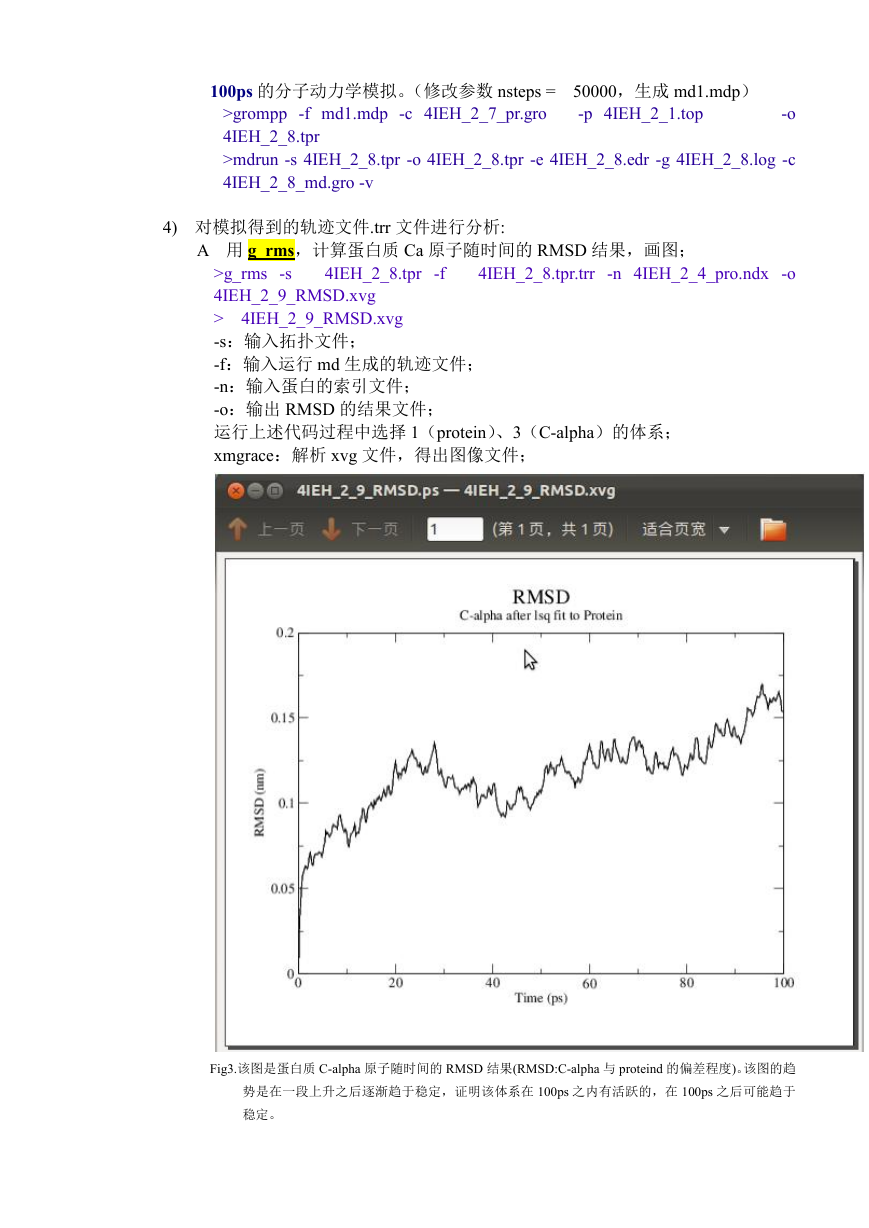
Fig3.该图是蛋白质 C-alpha 原子随时间的 RMSD 结果(RMSD:C-alpha 与 proteind 的偏差程度)。该图的趋
势是在一段上升之后逐渐趋于稳定,证明该体系在 100ps 之内有活跃的,在 100ps 之后可能趋于
稳定。
�
B 用 g_rmsf,计算 Ca 原子的 RMSF 变化幅度并画图
>g_rmsf -s 4IEH_2_8.tpr -f 4IEH_2_8.tpr.trr –o 4IEH_2_10_RMSD.xvg
>xmgrace 4IEH_2_10_RMSD.xvg
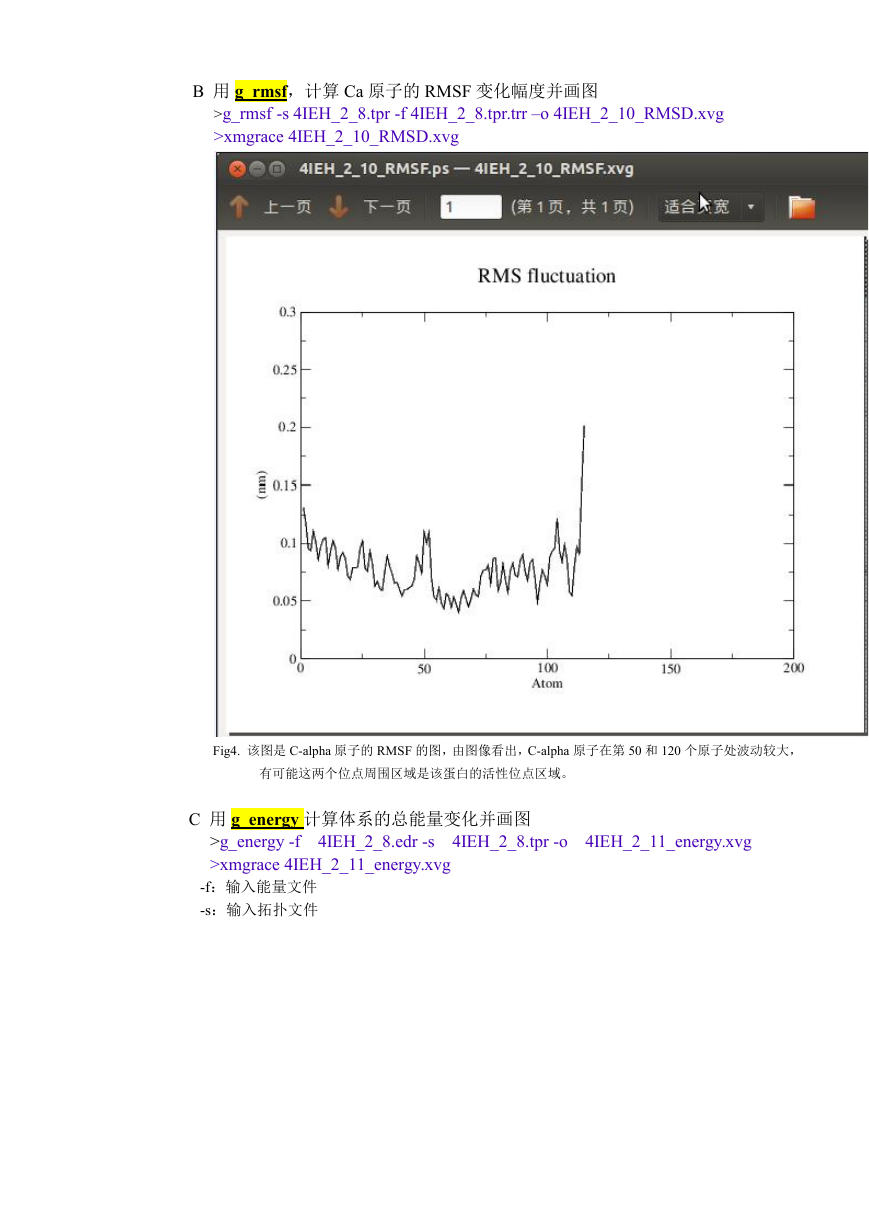
Fig4. 该图是 C-alpha 原子的 RMSF 的图,由图像看出,C-alpha 原子在第 50 和 120 个原子处波动较大,
有可能这两个位点周围区域是该蛋白的活性位点区域。
C 用 g_energy 计算体系的总能量变化并画图
4IEH_2_8.edr -s
>g_energy -f
>xmgrace 4IEH_2_11_energy.xvg
-f:输入能量文件
-s:输入拓扑文件
4IEH_2_8.tpr -o
4IEH_2_11_energy.xvg
�
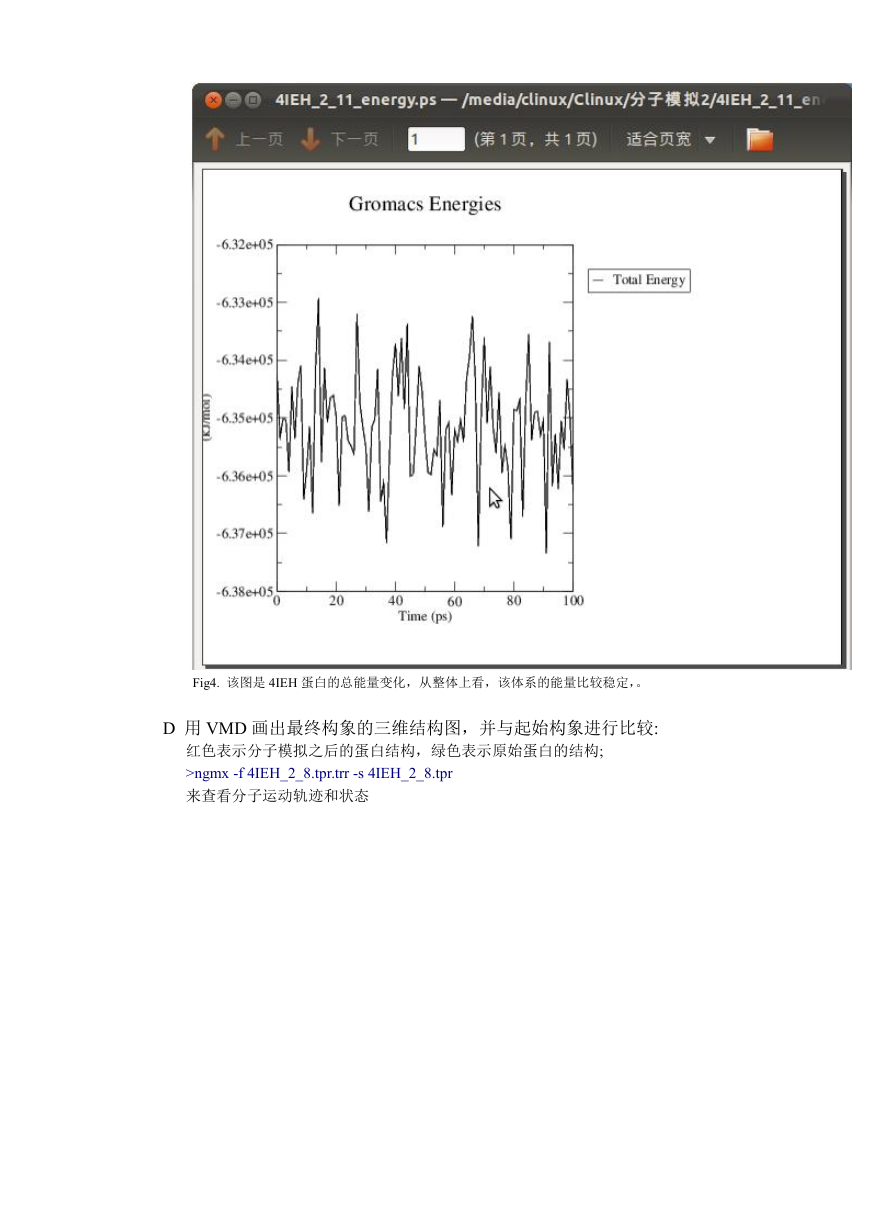
Fig4. 该图是 4IEH 蛋白的总能量变化,从整体上看,该体系的能量比较稳定,。
D 用 VMD 画出最终构象的三维结构图,并与起始构象进行比较:
红色表示分子模拟之后的蛋白结构,绿色表示原始蛋白的结构;
>ngmx -f 4IEH_2_8.tpr.trr -s 4IEH_2_8.tpr
来查看分子运动轨迹和状态
�












 2023年江西萍乡中考道德与法治真题及答案.doc
2023年江西萍乡中考道德与法治真题及答案.doc 2012年重庆南川中考生物真题及答案.doc
2012年重庆南川中考生物真题及答案.doc 2013年江西师范大学地理学综合及文艺理论基础考研真题.doc
2013年江西师范大学地理学综合及文艺理论基础考研真题.doc 2020年四川甘孜小升初语文真题及答案I卷.doc
2020年四川甘孜小升初语文真题及答案I卷.doc 2020年注册岩土工程师专业基础考试真题及答案.doc
2020年注册岩土工程师专业基础考试真题及答案.doc 2023-2024学年福建省厦门市九年级上学期数学月考试题及答案.doc
2023-2024学年福建省厦门市九年级上学期数学月考试题及答案.doc 2021-2022学年辽宁省沈阳市大东区九年级上学期语文期末试题及答案.doc
2021-2022学年辽宁省沈阳市大东区九年级上学期语文期末试题及答案.doc 2022-2023学年北京东城区初三第一学期物理期末试卷及答案.doc
2022-2023学年北京东城区初三第一学期物理期末试卷及答案.doc 2018上半年江西教师资格初中地理学科知识与教学能力真题及答案.doc
2018上半年江西教师资格初中地理学科知识与教学能力真题及答案.doc 2012年河北国家公务员申论考试真题及答案-省级.doc
2012年河北国家公务员申论考试真题及答案-省级.doc 2020-2021学年江苏省扬州市江都区邵樊片九年级上学期数学第一次质量检测试题及答案.doc
2020-2021学年江苏省扬州市江都区邵樊片九年级上学期数学第一次质量检测试题及答案.doc 2022下半年黑龙江教师资格证中学综合素质真题及答案.doc
2022下半年黑龙江教师资格证中学综合素质真题及答案.doc