- 1 -
目录
第 1 章 前 言........................................................................................................................................ 2
第 2 章 使用环境要求..........................................................................................................................3
2.1
JAVA 虚拟机环境......................................................................................................................3
2.2 系统环境要求.......................................................................................................................... 3
第 3 章 软件架构.................................................................................................................................. 5
第 4 章 软件的使用.............................................................................................................................. 6
4.1 软件运行界面.......................................................................................................................... 6
4.2 软件菜单功能介绍.................................................................................................................. 7
4.3 系统数据信息的读取.............................................................................................................. 8
4.4 系统数据信息分析................................................................................................................ 15
4.5 系统可视化展示功能描述.................................................................................................... 15
4.6 系统数据信息分析结果保存................................................................................................ 18
4.7 系统数据信息分析结果评估................................................................................................ 19
4.8 系统数据信息分析算法的有效性检验................................................................................ 38
4.9 系统蛋白质功能注释信息查看............................................................................................ 40
4.10 系统中插件信息查看............................................................................................................ 42
4.11 系统帮助信息查看................................................................................................................ 44
第 5 章 基于 CLUSTERE 系统插件的开发.................................................................................... 47
5.1 插件开发接口介绍................................................................................................................ 47
5.2 数据信息分析算法类插件开发............................................................................................ 49
5.3 分析结果评估方法类插件开发............................................................................................ 57
MEC 模型.......................................................................................................错误!未定义书签。
WLMF/GC 模型............................................................................................. 错误!未定义书签。
5.4 下载版的使用........................................................................................ 错误!未定义书签。
5.5
WEB 版的使用...................................................................................... 错误!未定义书签。
第 6 章 输出结果格式........................................................................................ 错误!未定义书签。
第 7 章 常见问题................................................................................................................................63
�
- 2 -
第 1 章 前 言
蛋白质网络聚类算法分析平台 ClusterE1.0 是中南大学信息科学与工程学院生物信息研
究组独立开发的生物信息分析平台。
蛋白质网络聚类是识别功能模块的重要手段,不仅有利于理解生物系统的组织结构,对
预测蛋白质功能也具有重要的意义。针对目前蛋白质网络聚类算法缺乏有效分析软件的事
实,本项目设计并实现了一个新的蛋白质网络聚类算法分析平台 ClusterE。该平台实现了查
全率、查准率、敏感性、特异性、功能富集分析等聚类评估方法,并且集成了 FAG-EC、Dpclus、
Monet、IPC-MCE、IPCA 等聚类算法,不仅可以对蛋白质网络聚类分析结果进行可视化,
并且可以在不同聚类分析指标下对多个聚类算法进行可视化比较与分析。该平台具有良好的
扩展性,其中聚类算法以及聚类评估方法都是以插件形式集成到系统中。
�
- 3 -
第 2 章 使用环境要求
为了提供本平台的可移植性 ClusterE 系统采用了 java 语言实现。因此只需要在电脑中
任何系统下装上虚拟机 JDK,便可以方便的运行本软件。同时为了解决部分用户安装 JDK 的
麻烦,本平台提供了两种版本:胖客户端以及瘦客户端。顾名思义,在此胖客户端指的是已
经集成了 JDK 环境在内的平台,其优点是无需去配置 java 环境,缺点是客户端系统占空间
比较大。而瘦客户端指的是没有集成 JDK 环境在内的平台,其优点是占空间比较小,但缺
点是需要配置 java 环境。一下将介绍 java 虚拟机环境的搭建以及本平台对系统环境要求。
2.1 Java 虚拟机环境
由于 java 虚拟机 JDK 版本较多,并且某些本版之间变化较大,因此 JDK 版本要求 1.5
及以上版本。
JDK 下 载 : 首 先 去 http://java.sun.com/javase/downloads/index.jsp 下 载 最 新 版 本
JDK1.6 。 一 切 下 载 后 选 择 安 装 路 径 , 例 如 我 选 择 安 装 在 "c:\Program
Files\Java\jdk1.6.0_02",安装到计算机上,这可能会花费数分钟时间,这与系统
性能有关。
JDK 配置:右击“我的电脑”,打开“属性”,选择“高级”里面的“环境变量”。
在新打开的界面中系统变量需要设置三个属性。在没安装过 JDK 的电脑中 path
属性是本来存在的。(1)点击“新建”,然后变量名写上"java_home",顾名思义
就 是 java 的 安 装 路 径 , 然 后 在 变 量 值 写 上 刚 才 的 安 装 路 径 "c:\Program
Files\Java\jdk1.6.0_02"。(2)在系统变量里找到 path,点击编辑。path 的含义就是
系 统 在 任 何 路 径 下 都 可 以 识 别 java 命 令 。 添 加 变 量 值
";%java_home%\bin;%java_home%\jre\bin"。(其中"%java_home%"的意思是刚才设
置的 java_home 的值)(3)点击“新建”,然后变量名写上"classpath",该变量的
含义为 java 加载类(bin or lib)的路径,只有类在 classpath 中,java 命令才能识别。
其值为".;%java_home%\lib;%java_home%\lib\tools.jar"(要加圆点.表示当前路径)。
JDK 环境验证:点击“开始”->“运行”,输入"cmd",进入命令行界面,打入"java
java version "1.6.0_02"(不同版本号则
-version",如果安装成功,则系统显示
不同)。
2.2 系统环境要求
本系统是一个开源的可移植平台,可在任何装有 JDK 的操作系统中运行。具体的使用
�
- 4 -
环境要求如下:
操作系统:无要求;
编译环境:JDK1.5 及以上;
处理器和内存的要求由所需要处理的数据量大小决定。
综上,可以看出 ClusterE 对系统使用环境要求低,其可移植性高,使用方便,能够适应
各种使用环境。
�
- 5 -
第 3 章 软件架构
对于使用环境来说,蛋白质网络聚类算法分析平台 ClusterE 系统是一个移植性高的平
台,但是对于系统本身设计来说它也是一个开源的非常优秀的可扩展平台:它能够非常方便
的添加新的聚类算法,以及新的聚类评估方法。任何使用本平台的用户都能方便的将聚类算
法以及评估方法以插件的方式集成到本平台中来。
图 3-1 ClusterE 的基本架构
�
- 6 -
第 4 章 软件的使用
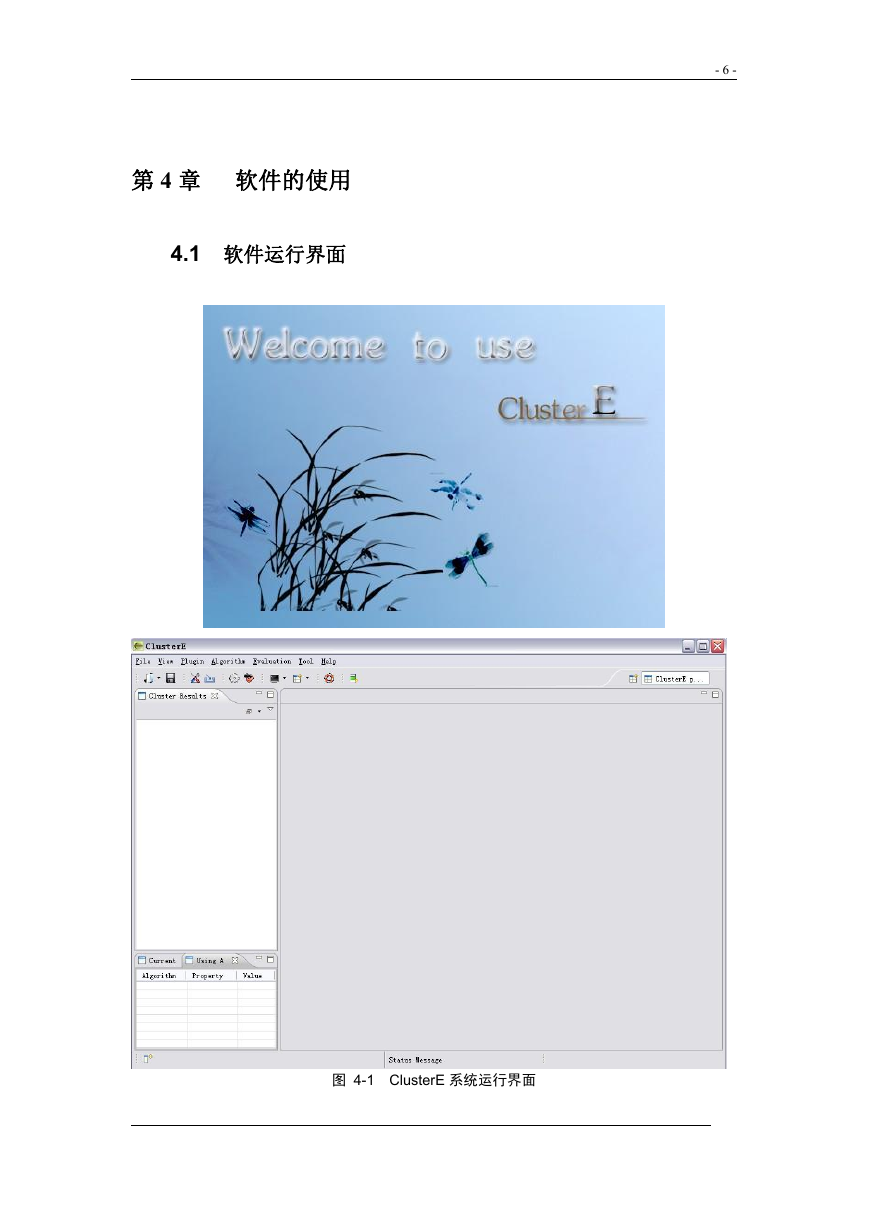
4.1 软件运行界面
图 4-1 ClusterE 系统运行界面
�
- 7 -
4.2 软件菜单功能介绍
File->Open->Text file:系统打开蛋白质相互作用网络数据并且读取。
File->Open->Clusters:系统打开蛋白质网络聚类分析算法已经实现的分析结果。
File-> Save:系统保存蛋白质网络聚类算法分析结果,该保存分为两种保存方式:
简易保存(不包含原始蛋白质网络信息)以及完整保存(包含原始蛋白质网络信息)。
File->Exit:退出系统。
View->Show View->Cluster Result view:系统打开蛋白质网络聚类算法分析结果
展示(以树形方式,每一个节点表示一个功能模块)视图。
View->Show View->Current network:系统打开当前完整蛋白质相互作用网络的基
本信息展示视图。其中基本信息包括节点数、边数以及平均度。
View->Show View->Using algorithms:系统打开当前所有聚类算法分析结果信息
展示视图。该信息包括,算法识别的功能模块数、最大模块规模、最小模块规模
以及平均度。
View->Show Editor->Cluster Editor:系统打开展示蛋白质网络聚类算法分析结果
的编辑器。每当点击 Cluster Result view 视图中的一个树节点在该编辑器中则相
应的以网络图方式可视化展示该功能模块,其中每个蛋白质以圆形节点表示,而
蛋白质间的相互作用以边的形式表示,网络图可以完整拖动,也可以拖动单个节
点。
View->Show Editor->Network Editor:系统打开展示当前完整蛋白质相互作用网络
图的编辑器,同样以网络图方式可视化展示蛋白质间的相互作用。
View->Show Editor->Evaluation Editor:系统打开展示蛋白质网络聚类算法评估结
果的编辑器。
View->Show Editor->Open Perspective:系统打开默认是视图。
View->Show Editor->Other:系统打开视图列表对话框。用户可以选择相应视图打
开。
Plugin->Plugin Manage:系统打开显示当前系统中已有插件列表对话框。用户可
以查看各个插件的详细信息。
Plugin->Import Plugin:系统导入新的插件到系统插件目录下。
Algorithm:该菜单下显示当前系统中所有蛋白质网络聚类算法,该菜单下子菜单
都是根据系统中已有插件动态生成。本系统中现以有算法:FAG-EC、DPclus、
Monet、HC-PIN、K-means、IPC-MCE。
Evaluation->Clustering Results:系统打开显示当前所有聚类算法分析结果基本信
息的对话框。
Evaluation :该菜单下显示当前系统中所有的蛋白质聚类结果评估方法,该菜单
下子菜单都是根据系统中已有插件动态生成。本系统目前已有的评估方法有:
Basic-Information(基本信息评估)、C-Score(其中包括功能富集程度评估 p-value、
�
- 8 -
查准率评估 precision、查全率评估 recall、综合评估 f-measure)、Comparision with
known complexes( 其 中 包 含 与 已 知 蛋 白 质 复 合 物 匹 配 评 估 、 敏 感 性 评 估
Sensitivity、特异性评估 Speicitivity)。
Tool->Paramater Set:系统打开设置系统参数对话框。其中主要包括系统基本信息
设置(画图前景色、背景色、特殊节点颜色、节点大小、线的宽度、是否显示节点、
是否显示节点标签、是否显示边等信息设置),以及蛋白质功能注释信息设置(本
系统集成了来自 MIPS,GO 数据库的蛋白质功能注释信息)。
Tool ->Gene Annotation:系统打开蛋白质功能注释信息对话框,在此对话框中可
以查看来自 MIPS,GO 数据库的蛋白质功能注释信息,也可以查询当前系统读入
蛋白质网络中各个蛋白质的功能注释信息并附加有搜索功能。当然用户也可以读
入系统新的蛋白质网络数据来查看该蛋白质网络中各个蛋白质的功能注释信息。
Tool->Change Network:系统打开随机改变当前蛋白质相互作用网络信息对话框,
在此对话框中可以设置获取随机蛋白质相互作用网络、获取原始蛋白质网络、随
机减少原始蛋白质网络中的边数、随机增加原始蛋白质网络中的边数。这样设计
的目的是为了将原始蛋白质网络聚类结果与随机蛋白质网络聚类结果进行对比,
从而去验证聚类算法的有效性。
Help->About:系统打开显示本平台基本信息对话框。
Help->Help content:系统打开平台帮助文档,该文档中包含对该平台基本操作演
示,基于该平台插件开发,以及实现该平台的所有 API 文档。
4.3 系统数据信息的读取
蛋白质网络相互作用信息读取
选择菜单 File->Open->Text File,或者工具栏
下拉菜单中 Text File,读取蛋白质
相互作用网络,即 Protein-protein Interaction Network(PPI 网络),数据格式如下图 4-1
所示。
�















 2023年江西萍乡中考道德与法治真题及答案.doc
2023年江西萍乡中考道德与法治真题及答案.doc 2012年重庆南川中考生物真题及答案.doc
2012年重庆南川中考生物真题及答案.doc 2013年江西师范大学地理学综合及文艺理论基础考研真题.doc
2013年江西师范大学地理学综合及文艺理论基础考研真题.doc 2020年四川甘孜小升初语文真题及答案I卷.doc
2020年四川甘孜小升初语文真题及答案I卷.doc 2020年注册岩土工程师专业基础考试真题及答案.doc
2020年注册岩土工程师专业基础考试真题及答案.doc 2023-2024学年福建省厦门市九年级上学期数学月考试题及答案.doc
2023-2024学年福建省厦门市九年级上学期数学月考试题及答案.doc 2021-2022学年辽宁省沈阳市大东区九年级上学期语文期末试题及答案.doc
2021-2022学年辽宁省沈阳市大东区九年级上学期语文期末试题及答案.doc 2022-2023学年北京东城区初三第一学期物理期末试卷及答案.doc
2022-2023学年北京东城区初三第一学期物理期末试卷及答案.doc 2018上半年江西教师资格初中地理学科知识与教学能力真题及答案.doc
2018上半年江西教师资格初中地理学科知识与教学能力真题及答案.doc 2012年河北国家公务员申论考试真题及答案-省级.doc
2012年河北国家公务员申论考试真题及答案-省级.doc 2020-2021学年江苏省扬州市江都区邵樊片九年级上学期数学第一次质量检测试题及答案.doc
2020-2021学年江苏省扬州市江都区邵樊片九年级上学期数学第一次质量检测试题及答案.doc 2022下半年黑龙江教师资格证中学综合素质真题及答案.doc
2022下半年黑龙江教师资格证中学综合素质真题及答案.doc