使用 FSL和 TrackVis分析 DTI数据
Alex / 2018-05-21 / free-learner@163.com
弥散加权成像(Diffusion Weighted Imaging, DWI)通过测量水分子的弥散程度来反映水分子所处的
组织结构特点,弥散张量成像(Diffusion Tensor Imaing, DTI)通过张量模型来定量地刻画水分子的弥
散程度,也是最简单的一种模型。作为初学者,总结一下使用 FSL和 TrackVis进行 DTI数据分析的基
本方法,包括涡流/头动校正(eddy current/head motion correction)、张量拟合(tensor fitting)和确
定性纤维束追踪(deterministic tractography)。
一、原始数据准备
(1)将从磁共振扫描仪上采集的原始数据转换成 NIFTI格式,可以使用 dcm2nii 或者 MRIConvert;
(2)假设转换成 NIFTI格式后的数据名为 dti_data.nii.gz,此外还会生成两个文本文件,分别表示磁场
梯度施加的强度和方向,这里假设这两个文件分别命名为 bvals和 bvecs。这两个文本文件的内容如下:
bvals:
bvecs:
(3)bvals和 bvecs这两个文件表示什么意思?DWI的大致原理是在不同方向上施加一个梯度磁场,这
个梯度磁场的强度用 b-value来表示,b-value越大、水分子弥散的距离越大、图像信号变化越明显;
DTI模型总共需要估计六个参数,所以至少需要在 6个不同方向上施加梯度,梯度方向用 b-vector来表
示;还需要至少一个没有施加梯度的图像作为参考,常称为 B0像。上面的 bvals文件总共 1行 65列,
表示总共采集了 65个图像,第 1个图像的 b-value是 0,后 64个图像的 b-value是 1000;bvecs文件
总共有 3行 65列,表示在每个图像上所施加的梯度方向。
�
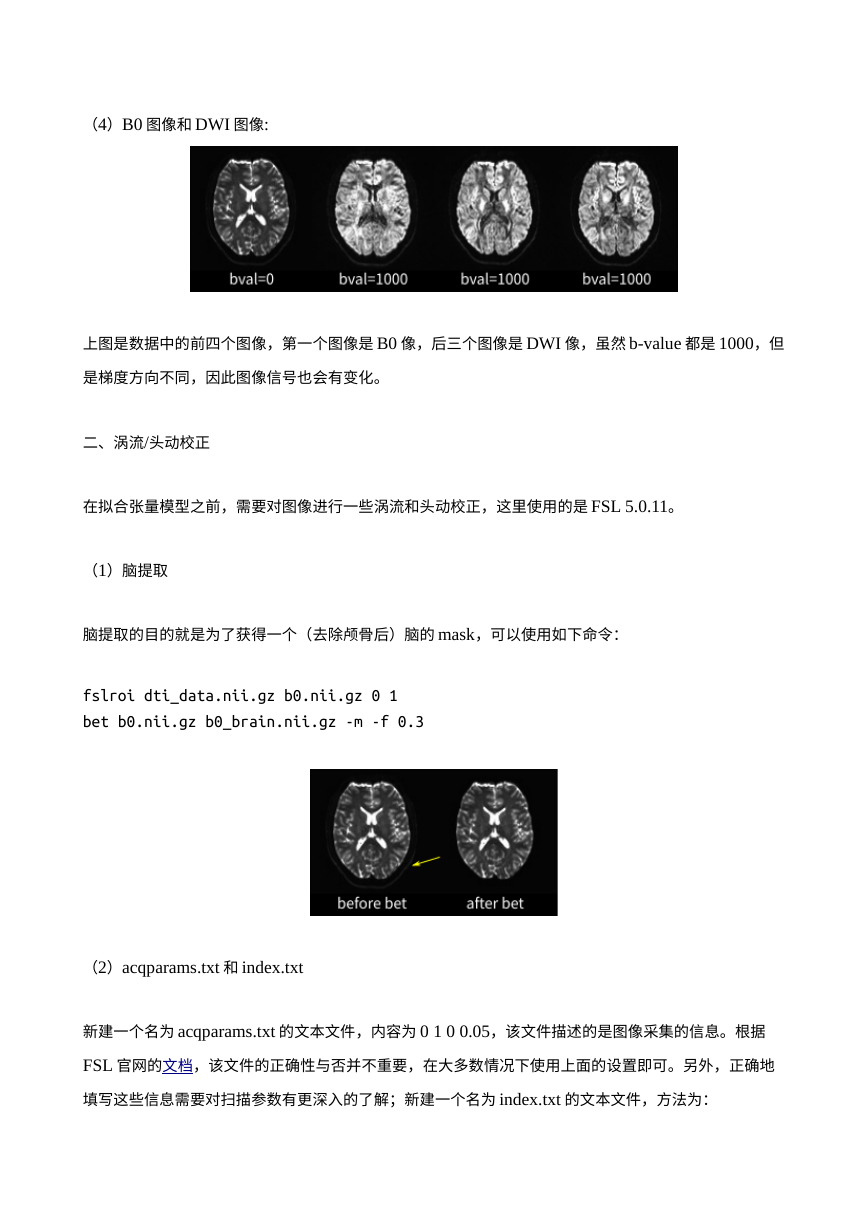
(4)B0图像和 DWI图像:
上图是数据中的前四个图像,第一个图像是 B0像,后三个图像是 DWI像,虽然 b-value都是 1000,但
是梯度方向不同,因此图像信号也会有变化。
二、涡流/头动校正
在拟合张量模型之前,需要对图像进行一些涡流和头动校正,这里使用的是 FSL 5.0.11。
(1)脑提取
脑提取的目的就是为了获得一个(去除颅骨后)脑的 mask,可以使用如下命令:
fslroi dti_data.nii.gz b0.nii.gz 0 1
bet b0.nii.gz b0_brain.nii.gz -m -f 0.3
(2)acqparams.txt和 index.txt
新建一个名为 acqparams.txt的文本文件,内容为 0 1 0 0.05,该文件描述的是图像采集的信息。根据
FSL官网的文档,该文件的正确性与否并不重要,在大多数情况下使用上面的设置即可。另外,正确地
填写这些信息需要对扫描参数有更深入的了解;新建一个名为 index.txt的文本文件,方法为:
�
indx=""
for ((i=1; i<=65; i+=1))
do
indx="$indx 1";
done
echo $indx > index.txt
上述代码的作用就是新建一个一列全为 1的数,并保存到 index.txt文件中;这个文件中的 1表示该图像
采集的参数对应于 acqparams.txt文件的第一行。由于 acqparams.txt里只有一行,所以所有图像都是
1。
(3)涡流/头动校正
eddy_openmp --imain=dti_data.nii.gz --mask=b0_brain_mask.nii.gz \
--acqp=acqparams.txt --index=index.txt --bvecs=bvecs --bvals=bvals \
--out=eddy_corrected_data
eddy_corrected_data.nii.gz即为涡流/头动校正后的数据,eddy_corrected_data.eddy_rotated_bvecs
为头动校正后的 b-vector文件。
三、张量拟合
dtifit -k eddy_corrected_data.nii.gz -o dti -m b0_brain_mask.nii.gz \
-r eddy_corrected_data.eddy_rotated_bvecs -b bvals --save_tensor
张量拟合结束后就可以得到一些定量指标,比如 FA(Fractional Anisotropy)和 MD(Mean
Diffusivity):
�
FA反映的是组织结构的方向性,如果水分子的弥散运动在某些方向上受到阻碍,在另一些方向上不受阻
碍,则 FA较大;FA较大的地方主要是在白质部分。MD表征水分子在所有方向上的平均弥散距离,如
果没有受到阻碍,则 MD较大;MD较大的地方主要在脑脊液。Color FA的计算方法是第一个特征向量
(first eigenvector, 文件名为 dti_V1.nii.gz)乘上 FA;第一个特征向量被认为反映(一个体素内)纤维
束的平均方向,所以 Color FA表示 FA较大的区域的纤维束的方向。一般地,红色表示左右(X轴),
绿色表示前后(Y轴),蓝色表示上下(Z轴)。
四、确定性纤维束追踪
纤维束追踪就是重建出神经纤维束,分为确定性纤维束追踪和概率纤维束追踪两种类型,纤维束追踪需
要对人脑的纤维束有相当的先验知识(而我没有这样的先验知识)。这里使用 TrackVis进行确定性纤维
束追踪。由于纤维束追踪是根据张量拟合的结果进行的,我现在还不清楚是否可以用 FSL的拟合结果来
作为 TrackVis的输入,因此需要用 TrackVis重新进行张量拟合并在此基础上进行确定性纤维束追踪。
TrackVis 实际上包含两个相对独立的软件,一个是 TrackVis用于可视化,一个是 Diffusion Toolkit用
于张量拟合和纤维束追踪,这里不做区分。
(1)准备文件
由于 TrackVis不能进行涡流/头动校正,使用 FSL的校正后的结果作为输入。另外,TrackVis的 b-
vector的格式不同于 FSL,首先 FSL的 b-vector的格式是每列表示一个梯度方向,TrackVis是每行表
示一个梯度方向,其次 TrackVis需要去掉 b-value为 0所对应的梯度方向。下面是一个简单的 R脚本,
将 FSL的 bvecs转换为 TrackVis的格式:
tmp <- as.matrix(read.table(‘eddy_corrected_data.eddy_rotated_bvecs’));
tmp <- t(tmp); ## transpose the matrix
tmp <- tmp[-1,]; ## remove the b0 rows
write.table(tmp, ‘trackvis_bvecs’, row.names=FALSE, col.names=FALSE);
�
(2)张量拟合
上图即为修改后的 bvecs
dti_recon "eddy_corrected_data.nii.gz" "trackvis" -gm "trackvis_bvecs" \
-b 1000 -b0 1 -ot nii.gz
gm选项指定 b-vector文件,b选项表示 b-value,b0选项表示有多少个 b0图像。FSL和 TrackVis的
张量拟合结果有微小的差异(大约小数点后第三位)。
(3)全脑纤维束追踪
dti_tracker "trackvis" "tmp.trk" -at 35 -m "b0_brain_mask.nii.gz" \
-m2 trackvis_fa.nii.gz 0.2 -sl -it nii.gz
spline_filter "tmp.trk" 1 "trackvis.trk"
m选项表示将追踪限制在脑 mask内,m2选项表示将追踪限制在 FA大于 0.2的区域;spline_filter对
追踪结果做一些平滑。在命令行输入 trackvis trackvis.trk,即可查看追踪结果:
�
(4)基于 ROI纤维束追踪
上图是全脑的纤维束追踪结果,对于纤维束追踪的原理我现在还是模糊的,我当前的理解是,以每一个
体素为起点(seed),都可以得到一条重建的纤维,那么以所有体素为起点的结果叠加在一起即为全脑
的追踪结果。如果只想看到通过特定 ROI的纤维束,可以使用该 ROI作为过滤器,去除不感兴趣的纤维
束。这里假设我感兴趣的区域是大脑白质,我通过对 T1加权像进行分割和配准得到白质的 mask,即白
质 ROI,实现代码如下:
## 去除 T1像的颅骨
## 将 T1像分割成灰质、白质和脑脊液三类
bet t1.nii.gz t1_brain.nii.gz
fast -t 1 -n 3 -H 0.1 -I 4 -l 20.0 -o t1 t1_brain.nii.gz
fslmaths t1_pve_2.nii.gz -thr 0.5 -bin wm_mask.nii.gz
## 使用 BBR配准,将 B0像配准到 T1像
flirt -ref t1_brain.nii.gz -in b0_brain.nii.gz -dof 6 -omat b02t1_init.mat
flirt -ref t1_brain.nii.gz -in b0_brain.nii.gz -dof 6 -cost bbr \
-wmseg wm_mask.nii.gz -init b02t1_init.mat -omat b02t1.mat -out b02t1Warp
## 将 T1的白质 mask转换到 B0像空间
convert_xfm -omat t12b0.mat -inverse b02t1.mat
flirt -in wm_mask.nii.gz -ref b0_brain.nii.gz -out wm_maskWarp -init t12b0.mat \
-applyxfm -interp nearestneighbour
## 将白质 mask缩小,减少配准误差的影响
fslmaths wm_maskWarp -ero wm_mask_ero
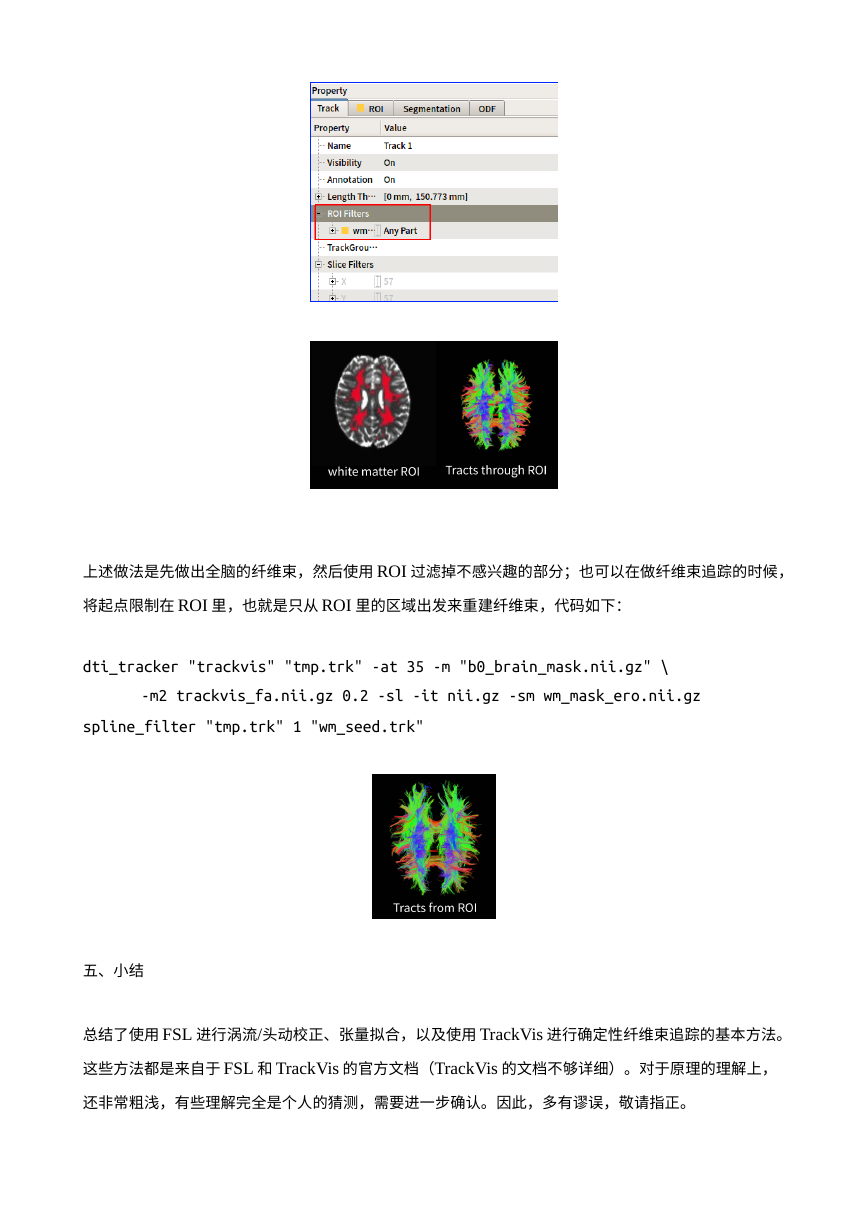
在 TrackVis的菜单栏选项 ROI -> New ROI From Nifti/Analyze Image,选择上一步得到的白质 ROI
mask,即 wm_mask_ero.nii.gz;在 TrackVis的右侧的 Property界面,选择 ROI Filters -> Toggle
Existing ROI,即得到通过白质 ROI的纤维束:
�
上述做法是先做出全脑的纤维束,然后使用 ROI过滤掉不感兴趣的部分;也可以在做纤维束追踪的时候,
将起点限制在 ROI里,也就是只从 ROI里的区域出发来重建纤维束,代码如下:
dti_tracker "trackvis" "tmp.trk" -at 35 -m "b0_brain_mask.nii.gz" \
-m2 trackvis_fa.nii.gz 0.2 -sl -it nii.gz -sm wm_mask_ero.nii.gz
spline_filter "tmp.trk" 1 "wm_seed.trk"
五、小结
总结了使用 FSL进行涡流/头动校正、张量拟合,以及使用 TrackVis进行确定性纤维束追踪的基本方法。
这些方法都是来自于 FSL和 TrackVis的官方文档(TrackVis的文档不够详细)。对于原理的理解上,
还非常粗浅,有些理解完全是个人的猜测,需要进一步确认。因此,多有谬误,敬请指正。
�












 2023年江西萍乡中考道德与法治真题及答案.doc
2023年江西萍乡中考道德与法治真题及答案.doc 2012年重庆南川中考生物真题及答案.doc
2012年重庆南川中考生物真题及答案.doc 2013年江西师范大学地理学综合及文艺理论基础考研真题.doc
2013年江西师范大学地理学综合及文艺理论基础考研真题.doc 2020年四川甘孜小升初语文真题及答案I卷.doc
2020年四川甘孜小升初语文真题及答案I卷.doc 2020年注册岩土工程师专业基础考试真题及答案.doc
2020年注册岩土工程师专业基础考试真题及答案.doc 2023-2024学年福建省厦门市九年级上学期数学月考试题及答案.doc
2023-2024学年福建省厦门市九年级上学期数学月考试题及答案.doc 2021-2022学年辽宁省沈阳市大东区九年级上学期语文期末试题及答案.doc
2021-2022学年辽宁省沈阳市大东区九年级上学期语文期末试题及答案.doc 2022-2023学年北京东城区初三第一学期物理期末试卷及答案.doc
2022-2023学年北京东城区初三第一学期物理期末试卷及答案.doc 2018上半年江西教师资格初中地理学科知识与教学能力真题及答案.doc
2018上半年江西教师资格初中地理学科知识与教学能力真题及答案.doc 2012年河北国家公务员申论考试真题及答案-省级.doc
2012年河北国家公务员申论考试真题及答案-省级.doc 2020-2021学年江苏省扬州市江都区邵樊片九年级上学期数学第一次质量检测试题及答案.doc
2020-2021学年江苏省扬州市江都区邵樊片九年级上学期数学第一次质量检测试题及答案.doc 2022下半年黑龙江教师资格证中学综合素质真题及答案.doc
2022下半年黑龙江教师资格证中学综合素质真题及答案.doc