PMF5.0 操作手册
1. 简介
1.1 手册目的
本手册的目的是方便 PMF5.0 软件使用者了解 PMF 所能实现的功能、软件的具
体操作方法,以快速实现 VOCs 的源解析工作。
1.2 名词定义
1.3 PMF5.0 的整体特征
用户可实现以下操作:
(1) 数据排序:单击 species 等数据列进行排序,单击 unsort 还原。
(2) 保存图片:界面上的所有图片,均可通过右击保存。因子成分谱和时间序
列还可以堆叠后再保存。
(3) 图片在新窗口显示:右键,单击 Floating Window,可在新窗口查看图片。
(4) 调节窗口大小:在浓度散点图窗口,可拉动 section 边框改变其长宽。
�
(5) 数据点信息显示:点击浓度散点图或时间序列图中的任意一点,最底下一
栏会显示这个点的所有信息。
(6) Help 文档:在每个单独窗口左下角都有一个 help 按钮,点击 help 可获得
当前窗口的一些解释和说明。
(7) 统计信息:例如散点图,在窗口底部有一栏会显示两个组分的统计信息。
2. 软件操作
2.1 数据准备
包含两部分:样品浓度矩阵和不确定性度矩阵。备注:数据如有更改需重新导入。
2.1.1 浓度数据
2.1.1.1 格式
PMF 运行首先需要给出一个样品浓度矩阵,软件支持.txt、.csv、.xls 、 .xlsx 格
式
2.1.1.2 排版
对于浓度数据,需要按照以下表格进行数据的排列和前准备工作。
第一列为:时间数据(可增加 Sample ID 列)
第一行:物种名称 第二行:单位(ug/m³)可加可不加
(1) 单站点浓度数据:例 csv 格式
(2)多站点同时解析,浓度数据:例 csv 格式
�
不允许出现空白值,若出现负值或数据缺失,则以其他值代替。后文将详述。
2.1.1.3 数据量
对于 PM2.5,要求不少于 10-20 个物种,超过 100 组数据。
对于 VOCs,则物种不少于 30 种,一般在 30-50 之间,数据量要达到 200 组。
2.1.2 不确定度
2.1.2.1 格式和排版
不确定性度涵盖了采样和分析误差等,可由实验室数据分析部门直接提供,若不
能则需自己计算。PMF5.0 允许两种不确定性的表达方式,一种是与浓度表格严
格对应的数据排列方式,只是不能包含单位。见下图:
另外一种是提供一个算式,见下图:
�
其中,第一行是物种名称,第二行是方法检出限 MDL,第三行是测量误差,本
例中为 3%。不确定性度不允许出现负值和 0。
2.1.2.2 不确定度计算(重要,对 PMF 运行结果影响很大)
(1)根据采样误差,测量误差,凭借经验估算不确定性度。
(2)根据测量误差和检出限计算不确定度
如果浓度值大于检出限 MDL,则
如果浓度值小于或等于 MDL,则
备注:低于检出限的数据用 1/2 MDL 来代替,其不确定度为 5/6 MDL。
缺失的数据用测量值的几何平均值代替,其不确定度值为 4 倍测量值的不确定
度值。用户可在数据导入界面指定一个缺失值的 indicator,如-999
此外:样本中单个物种“0”值超过该物种总样本数 80% 的不在模型中使用,
50% ~ 80%物种 UNC 扩大 3 倍,小于 50%的可用于受体模型。
2.2 数据导入和筛选
2.2.1 Data File 界面介绍
�
2.2.1.1
Input Files
依次导入浓度和不确定性数据,以.csv 或.txt 格式。设置 Missing Value Indicator
一栏,为 Replacing Missing Values with Species Median,也就是对于缺失值,不删
除整组数据,而是以中位数代替。其他全为默认设置,不需要改。
2.2.1.2 Output Files
在 EPA PMF\Output 后新建一个文件夹,保存输出结果,设置输出文件前缀,例
如 xp20160815 等,设置输出文件类型,可选择 EXCEL 07-10 工作簿(方便)。
当模型运行后,结果会保存再输出文件中。如果进行参数修改后重新运行,则需
要重新设置前缀,不然当前运行会将上次运行结果覆盖掉。结果文件及其包含的
内容如下:
*_base.xls – Profiles, Contributions, Residual, Run Comparison
*_diagnostics.xls – Summary, Input, Base Runs
当进行 bootstrapping 后,有以下输出文件:
① (*_profile_boot),
② DISP(*_DISPres1, *_DISPres2, *_DISPres3, *_DISPres4),
③ BS-DISP (*_BSDISP1, *_BSDISP2,*_BSDISP3, *_BSDISP4),
�
④ Fpeak (*_fpeak), and/or
⑤ constrained model runs (*_Constrained)
2.2.1.3 Configuration File
配置文件的作用是保存所有设置好的运行参数,方便下次调用。首次运行可不选
择配置文件,若想保存本次物种选择、剔除异常值、输入输出位置等操作,则可
在关闭界面前,保存配置。
至此,数据输入模块完成。
2.2.2 数据筛选方法(
)
2.2.2.1 数据筛选原则(设置 strong、weak、bad)
将物种分别设置 strong、weak、bad 意味着每类物种在参与最优解求解过程中其
重要性,例如,设置为 strong 则表示运算中该列数据权重较大,设置为 weak 权
重降低(不确定度增加 3 倍),设置为 bad 则不参与运算。
(1)S/N
样本中物种浓度信噪比 S/N 大于 1 为合格数据直接用于模型, 0. 5~ 1 物种的
通过扩大其不确定度( UNC) 降低计算权重,小于 0. 5 的不在模型中使用。
备注:之前 S/N 的算法是将所有浓度求和除以不确定性度之和,这种算法有很多缺陷。
如当有缺失值时不确定性度值增大,使 S/N 降低。新算法将浓度大于不确定性的数据纳入计
算,浓度小于不确定度的 dij=0。
(2)物种浓度
物种浓度较大的优先考虑设置为 strong,浓度特别低的物种,测量误差相对较大,
可酌情设置为 weak,或 bad。在 Concentration/Uncertainty 界面,系统自动生成每
个 species 的最小值,25th,50th,75th,最大值,通过对比 50th 可大概了解各物种浓度,
这部分也可辅助判断是否有异常值:0 或超大值。
(3)物种示踪意义
具有示踪意义的物种,即使其浓度较低,也可酌情设置为 strong。
设置后效果图见下图:
�
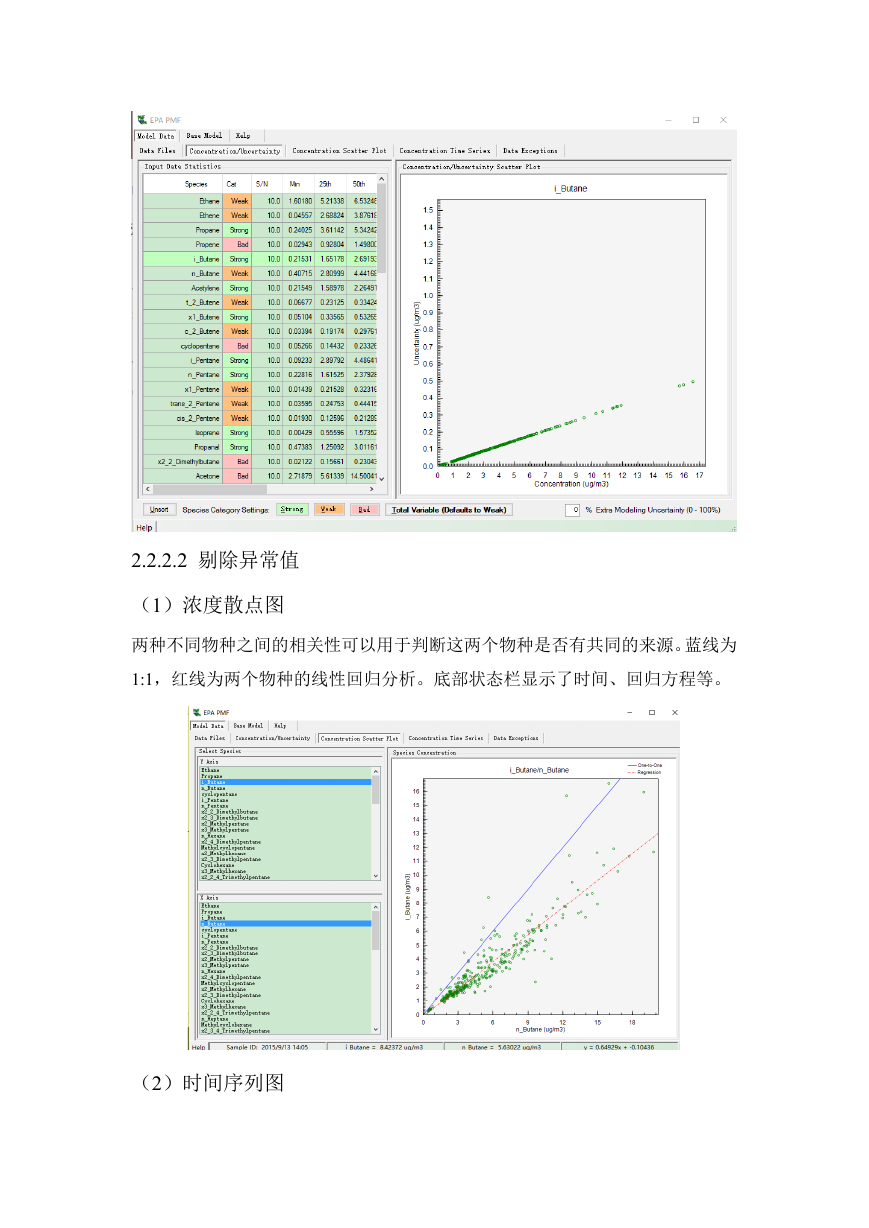
2.2.2.2 剔除异常值
(1)浓度散点图
两种不同物种之间的相关性可以用于判断这两个物种是否有共同的来源。蓝线为
1:1,红线为两个物种的线性回归分析。底部状态栏显示了时间、回归方程等。
(2)时间序列图
�
查看某物种的时间序列图是否有异常值,可与同期其他物种的时间序列图相互比
较,若有异常的极大值或者极小值,可将其排除。这是因为异常值的存在会影响
某一类源的常规特征的判断,引起解析结果的偏差。
选中要剔除的异常值,点击屏幕右下角的 exclude samples 删除,通过 restore
samples 可还原。
2.3 模型运转
2.3.1 因子数目选择
因子数即为解析的源的个数,可依据数据量大小和基于污染来源的初步判断从小
往大进行试算。一般从 4 算起,也就是说假设有 4 个源,看解析的结果是否可
靠。因子数选择得过少,则有些源分不开,因子数过多,则有些因子没意义。因
而需要不断改变因子数不断进行试算,才能得到最小和最稳定的 Q 值。
2.3.2 试算-其他设置
“Number of Runs” :每次运行的运算次数,一般默认20次,以找到最小的Q
值。可设为1-999之间的任意值。
“Number of Factors” :即因子数
“Seed” :运算的起点,可按系统默认随机选择起点。如果不勾选随机起点,改
为一固定值,则当下次运行启用相同的.cfg文件可以进行相同的运算得到相同的
结果。
设置好后,点击Base Model Run模块下的run,屏幕下方进度条即开始指示运算
过程,一般几分钟即可完成试算。
�














 2023年江西萍乡中考道德与法治真题及答案.doc
2023年江西萍乡中考道德与法治真题及答案.doc 2012年重庆南川中考生物真题及答案.doc
2012年重庆南川中考生物真题及答案.doc 2013年江西师范大学地理学综合及文艺理论基础考研真题.doc
2013年江西师范大学地理学综合及文艺理论基础考研真题.doc 2020年四川甘孜小升初语文真题及答案I卷.doc
2020年四川甘孜小升初语文真题及答案I卷.doc 2020年注册岩土工程师专业基础考试真题及答案.doc
2020年注册岩土工程师专业基础考试真题及答案.doc 2023-2024学年福建省厦门市九年级上学期数学月考试题及答案.doc
2023-2024学年福建省厦门市九年级上学期数学月考试题及答案.doc 2021-2022学年辽宁省沈阳市大东区九年级上学期语文期末试题及答案.doc
2021-2022学年辽宁省沈阳市大东区九年级上学期语文期末试题及答案.doc 2022-2023学年北京东城区初三第一学期物理期末试卷及答案.doc
2022-2023学年北京东城区初三第一学期物理期末试卷及答案.doc 2018上半年江西教师资格初中地理学科知识与教学能力真题及答案.doc
2018上半年江西教师资格初中地理学科知识与教学能力真题及答案.doc 2012年河北国家公务员申论考试真题及答案-省级.doc
2012年河北国家公务员申论考试真题及答案-省级.doc 2020-2021学年江苏省扬州市江都区邵樊片九年级上学期数学第一次质量检测试题及答案.doc
2020-2021学年江苏省扬州市江都区邵樊片九年级上学期数学第一次质量检测试题及答案.doc 2022下半年黑龙江教师资格证中学综合素质真题及答案.doc
2022下半年黑龙江教师资格证中学综合素质真题及答案.doc