基于Linux的生物信息操作环境
关键词:生物信息 操作环境
孔雷 高歌 吴健民 李哲 赵树起 唐汶 罗静初*
北京大学
20世纪90年代,人类基因组计划以及其
他模式生物基因组计划的实施和相继完成,使
核酸和蛋白质序列数据急速增长,以分子生
物信息为主要研究对象的生物信息学应运而
生。十多年来,生物信息学正逐步成为一门独
立学科,生物信息软件开发成了一个专门领
域。数据库搜索程序BLAST、多序列联配程序
Clustalw、系统发生分析软件Phylip以及大型序
列分析软件包EMBOSS等,已经成为分子生物
学研究中不可缺少的工具。公开、公共、公用
是生物信息软件的主要特征。这些软件通常采
用各种形式的开放源代码版权声明,以开源的
Linux操作系统为主要平台,多数由科研机构、
高等院校等学术部门自由开发,并提供给公众
使用。这是当前生物信息软件开发的主流。
然而,这些开源软件的开发,通常针对某
个研究领域的具体问题,以学术研究为主要目
的,以个人兴趣为主要动力,因此所编写的程
序往往缺乏详尽的文档和必要的用户支持。安
装和配置这类软件,需要具有一定计算机编程
及系统维护的相关知识和经验,多数生物学家
感到力不从心。即使是生物信息学家,乃至专
业系统管理员,面对如此众多的软件和有限的
文档,也会感到相当棘手。此外,随着生物信
息数据量激增和对数据深入研究,往往需要在
一个研究项目中同时使用多个软件。一个配置
良好、可提供各种生物软件的操作环境就显得
十分必要。GNU1项目生物学软件目录[1]下现有
15种软件,开放科学(The Open Science)项目
生物学软件目录[2]下有51种软件,绝大多数可
在Linux操作系统下运行。因此,以Linux为平
台开发这样一个操作环境,不仅必要,而且可
行。
通用Linux发行版软件仓库中的生物信息
软件多为RPM(Red Hat Package Manager,Red
Hat包管理器)格式,这些软件适合Red Hat/
Fedora等使用RPM软件包管理系统的Linux发行
版,如Bio-Linux[3]或BioRPM[4]。通用Linux发行
版Debian[5]和Gentoo[6]也提供数量较多的生物信
息学软件包,用户需要自行安装这些软件。而
基于Linux的生物信息操作环境则集成了大部分
生物信息软件,便于安装使用。生物信息操作
环境主要包括两类,一类是光盘型生物信息操
作环境,即整个系统集成后刻录在一张或几张
光盘上,运行时只需将光盘插入计算机,不需
要预先在硬盘上安装;另一类则需将集成后的
系统安装到硬盘上,使用时通过硬盘启动后进
入预先设定的操作环境。
光盘型生物信息操作环境
光盘型生物信息操作环境为LiveCD类型。
* 北京大学生物信息中心、北京大学蛋白质工程和植物基因工程国家重点实验室、北京大学生命科学
学院 北京 100871
1 GNU始于1984年,旨在发展一个类似Unix、且为自由软件的完整操作系统——GNU系统。GNU是由
“GNU's Not Unix”所递归定义的首字母缩写语,发音为“guh-NEW”。
51
第 2 卷 第 3 期 2006 年 5 月 �
封面报道
其特点是使用简单,适合不太熟悉Linux系统的
用户。使用时只需从装有LiveCD的系统光盘启
动,即可进入操作环境。由于光盘运行时只在
内存中建立临时文件系统,不用担心由于操作
不慎而破坏本地硬盘文件系统。这种操作环境
通常对计算机硬件环境要求较低,比较适合教
学和演示。其缺点是运行速度和数据处理能力
受内存大小限制,数据保存不够方便,一般使
用优盘(U-disk)保存。代表性软件有:
1. BioKnoppix[7] 是基于通用Knoppix LiveCD
LiveCD,目前版本为1.0,同样基于Knoppix,
提供20余种生物信息学软件,大多数有图形界
面,默认桌面管理器KDE。除生物信息学软件
外,还提供序列数据,如大肠杆菌和蓝细菌基
因组序列等。Vigyaan主页上也为初学者提供了
在线教程和演示。
3. DNALinux[9] 是GenesDigitales开发的光盘
型生物信息操作环境,目前稳定版本是0.42,
开发的Linux光盘型生物信息操作环境,目前
为Beta-0.21版。该系统以分子生物学家为主
要用户群,适合演示及分子生物学教学。最
新版BioKnoppix集成了10余种生物信息学软件
包,绝大多数有图形用户界面,如EMBOSS/
Jemboss、Artemis、ClustalX、Cn3D、ImageJ
及RasMol等。BioKnoppix默认的桌面管理器
是KDE(K Desktop Environment,K桌面系
统),风格类似微软视窗(W i n d o w s),便
于 大 多 数 习 惯 使 用 视 窗 的 生 物 学 家 入 门 使
用。BioKnoppix也提供了一些开发工具包,如
BioPython、BioPerl及BioConductor等,但在演
示和教学活动中,较少用到这些开发工具。
BioKnoppix在其主页上提供了使用简例和一个
可视化教程。
图为Vigyaan界面
该版本是基于Slax 4.1.4开发的。DNALinux提供
了约20种生物信息学软件,主要是关于核酸及
蛋白质序列处理工具,这些工具多数没有图形
用户界面。DNALinux提供部分BLAST格式的蛋
白质序列数据库。
硬盘安装型生物信息操作环境
硬盘安装型生物信息操作环境一般都基
于较为流行的Linux发行版,如Debian和Fedora
等。这类环境通常提供基于图形界面的安装程
序,便于具有一定Linux系统基础的用户安装。
这种Linux生物信息操作环境功能一般比光盘型
的系统全面,可作为生物学研究者日常桌面系
统。若安装到系统硬件配置较高的服务器上,
亦可作为多任务服务器。代表性软件有:
2. Vigyaan[8] 是以生物信息学家、计算生
物 学 家 和 计 算 化 学 家 为 目 标 用 户 群 开 发 的
1. Bio-Linux[10] 是由英国国家环境研究委员
会生物信息中心开发的一种硬盘安装型Linux生
52
第 2 卷 第 3 期 2006年 5 月 �
多人使用一台服务器。硬盘安装型Bio-Linux是
基于通用Linux发行版Debian环境,而国内广大
Linux用户,尤其是生物学家对Redhat/Fedora系
列和RPM包管理系统更为熟悉。为此,我们以
Fedora2为基础平台,开发了一个硬盘安装型的
Linux生物信息操作环境BioLand。
BioLand旨在为生物学家提供一个易于安装
维护的生物信息操作环境,并为中小型生物学
实验室或大型生物实验室中的工作组提供一个
可共享的数据平台和计算服务器。Fedora2是桌
面型RedHat的后继版,整合了大量Linux常用系
统软件,具有良好的用户基础。因此BioLand选
择Fedora Core Linux作为基础平台,采用RPM包
管理系统整合相关生物信息软件和数据库自动
更新程序。
BioLand当前版本是BioLand 2004,基于
Fedora Core 2,整合了BLAST、EMBOSS、
F A S T A 和 H M E E R 等 2 0 余 种 生 物 软 件 以 及
BLOCKS、PRINTS、PROSITE等蛋白质序列模
块数据库。BioLand的目标用户群中包括实验生
物学家,他们往往不熟悉命令行操作方式,因
物信操作环境,最新版4.0基于Debian Linux。
该操作环境提供50多个生物信息学软件包,包
括ClustalW和BLAST等常用软件,同时还提供
开发工具和详尽说明文档以及文档查询功能。
为方便使用者,Bio-Linux还在其桌面环境菜单
中加入了生物信息软件快捷方式。基于Debian
Linux使得该系统易于升级更新。如果作为服务
器使用,还可使用Bio-Linux自带防火墙和基于
Web的管理工具。
图为Bio-Linux及其包含的部分软件包
2. B i o B r e w [11] 是由
bioinfromatics.org组织开发
的一款专为机群系统定制
的Linux生物信息操作环
境,提供了一些生物学序
列联配软件。此外,上述
DNALinux也提供硬盘安
装型生物信息操作环境。
生 物 信 息 操 作
环境BioLand
上述两类Linux生物
信息操作环境各有利弊,
光盘型不需安装,使用简
单,但功能有限,不利于
2 Fedora是Red Hat桌面版本的延续。
53
第 2 卷 第 3 期 2006 年 5 月 �
封面报道
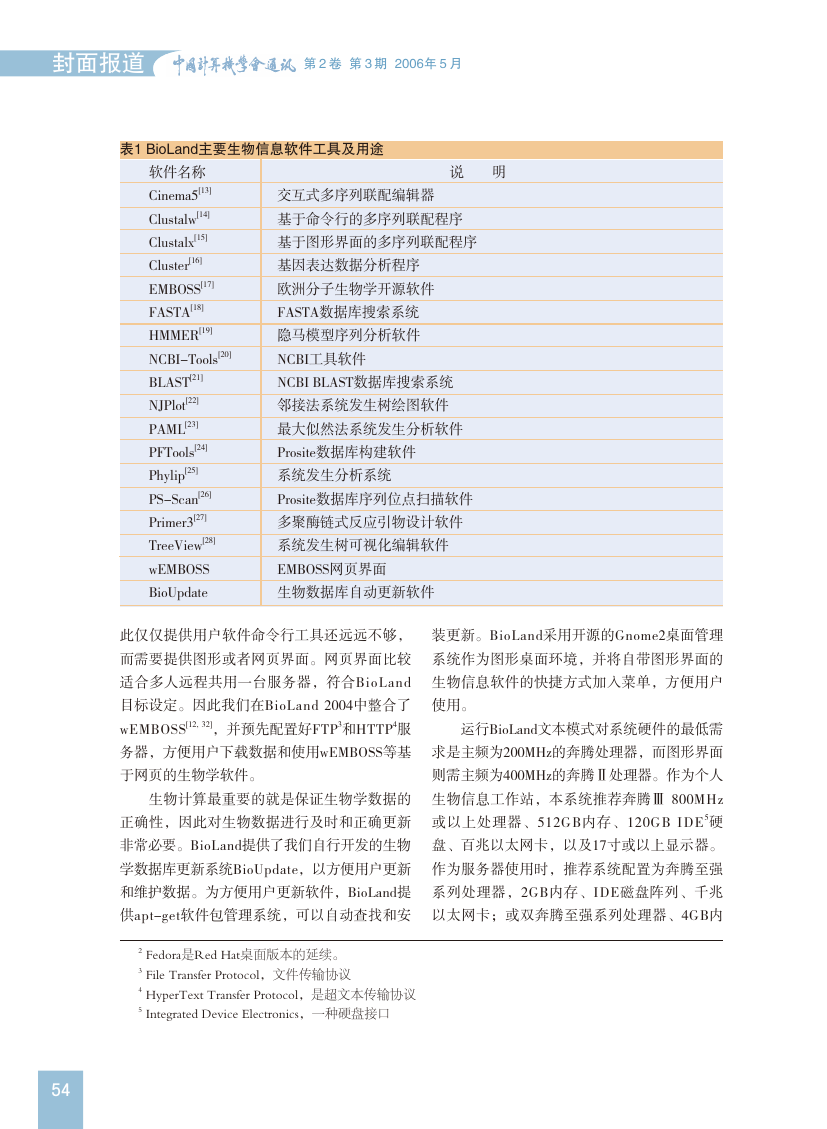
表1 BioLand主要生物信息软件工具及用途
说 明
交互式多序列联配编辑器
基于命令行的多序列联配程序
基于图形界面的多序列联配程序
基因表达数据分析程序
欧洲分子生物学开源软件
FASTA数据库搜索系统
隐马模型序列分析软件
软件名称
Cinema5[13]
Clustalw[14]
Clustalx[15]
Cluster[16]
EMBOSS[17]
FASTA[18]
HMMER[19]
NCBI-Tools[20]
BLAST[21]
NJPlot[22]
PAML[23]
PFTools[24]
Phylip[25]
PS-Scan[26]
Primer3[27]
TreeView[28] 系统发生树可视化编辑软件
wEMBOSS
BioUpdate
生物数据库自动更新软件
NCBI工具软件
NCBI BLAST数据库搜索系统
邻接法系统发生树绘图软件
最大似然法系统发生分析软件
Prosite数据库构建软件
系统发生分析系统
Prosite数据库序列位点扫描软件
多聚酶链式反应引物设计软件
EMBOSS网页界面
此仅仅提供用户软件命令行工具还远远不够,
而需要提供图形或者网页界面。网页界面比较
适合多人远程共用一台服务器,符合BioLand
目标设定。因此我们在BioLand 2004中整合了
wEMBOSS[12, 32],并预先配置好FTP3和HTTP4服
务器,方便用户下载数据和使用wEMBOSS等基
于网页的生物学软件。
生物计算最重要的就是保证生物学数据的
正确性,因此对生物数据进行及时和正确更新
非常必要。BioLand提供了我们自行开发的生物
学数据库更新系统BioUpdate,以方便用户更新
和维护数据。为方便用户更新软件,BioLand提
供apt-get软件包管理系统,可以自动查找和安
装更新。BioLand采用开源的Gnome2桌面管理
系统作为图形桌面环境,并将自带图形界面的
生物信息软件的快捷方式加入菜单,方便用户
使用。
运行BioLand文本模式对系统硬件的最低需
求是主频为200MHz的奔腾处理器,而图形界面
则需主频为400MHz的奔腾Ⅱ处理器。作为个人
生物信息工作站,本系统推荐奔腾Ⅲ 800MHz
或以上处理器、512GB内存、120GB IDE5硬
盘、百兆以太网卡,以及17寸或以上显示器。
作为服务器使用时,推荐系统配置为奔腾至强
系列处理器,2GB内存、IDE磁盘阵列、千兆
以太网卡;或双奔腾至强系列处理器、4GB内
2 Fedora是Red Hat桌面版本的延续。
3 File Transfer Protocol,文件传输协议
4 HyperText Transfer Protocol,是超文本传输协议
5 Integrated Device Electronics,一种硬盘接口
54
第 2 卷 第 3 期 2006年 5 月 �
存、SCSI6磁盘阵列、千兆以太网卡。
BioLand从开发至今,已被国内外用户大
量下载,被国外多家网站引用并在多家杂志刊
登[29,30,31]。我们用BioLand作为北京大学及中国
农业科学院研究生院开设的实用生物信息技术
课程的基本教学环境,取得了很好的效果。目
前,正在进一步扩充软件工具,包括蛋白质空
间结构显示软件、基因芯片数据分析软件等,
并增加常用生物信息数据库。
孔雷
北京大学生物信息中心博士研究生。
罗静初
北京大学生物信息中心教授。
参考文献和网址
[1] http://directory.fsf.org/science/biology/
[2] http://www.openscience.org/links/index.php?section=17
[3] http://www.biolinux.org/
[4] http://uberh4x0r.org/~yax/biorpm/
[5] http://www.debian.org/
[6] http://www.gentoo.org/
[7] http://bioknoppix.hpcf.upr.edu/
[8] http://www.vigyaancd.org/
[9] http://www.dnalinux.com/
[10] http://envgen.nox.ac.uk/biolinux.html
[11] http://bioinformatics.org/biobrew/
[12] http://www.wemboss.org/
[13] http://umber.sbs.man.ac.uk/dbbrowser/CINEMA2.1/
[14] http://www.ebi.ac.uk/clustalw/
[15] http://bips.u-strasbg.fr/fr/Documentation/ClustalX/
[16] http://bonsai.ims.u-tokyo.ac.jp/~mdehoon/software/cluster/
[17] http://emboss.sourceforge.net/
[18] http://fasta.bioch.virginia.edu/
[19] http://hmmer.wustl.edu/
[20] http://www.ncbi.nlm.nih.gov/Tools/
[21] http://www.ncbi.nlm.nih.gov/BLAST/
[22] http://pbil.univ-lyon1.fr/software/njplot.html
[23] http://abacus.gene.ucl.ac.uk/software/paml.html
[24] http://www.expasy.org/prosite/
[25] http://evolution.genetics.washington.edu/phylip.html
[26] http://www.expasy.org/prosite/
[27] http://frodo.wi.mit.edu/primer3/
[28] http://darwin.zoology.gla.ac.uk/~rpage/treeviewx/
[29] The Bioinformatics Playground, Tiwari, B and Field, D. Linux User and Developer. 2005. Issue 46. pp. 50-56
[30] http://gchelpdesk.ualberta.ca/news/17mar05/cbhd_news_17mar05.php
[31] http://gchelpdesk.ualberta.ca/news/03mar05/cbhd_news_03mar05.php#GearingUp
[32] wEMBOSS: a web interface for EMBOSS, Martín Sarachu and Marc Colet, Bioinformatics, Feb 2005; 21: 540
- 541
6 Small Computer System Interface,小型计算机系统接口
55
第 2 卷 第 3 期 2006 年 5 月 �









 2023年江西萍乡中考道德与法治真题及答案.doc
2023年江西萍乡中考道德与法治真题及答案.doc 2012年重庆南川中考生物真题及答案.doc
2012年重庆南川中考生物真题及答案.doc 2013年江西师范大学地理学综合及文艺理论基础考研真题.doc
2013年江西师范大学地理学综合及文艺理论基础考研真题.doc 2020年四川甘孜小升初语文真题及答案I卷.doc
2020年四川甘孜小升初语文真题及答案I卷.doc 2020年注册岩土工程师专业基础考试真题及答案.doc
2020年注册岩土工程师专业基础考试真题及答案.doc 2023-2024学年福建省厦门市九年级上学期数学月考试题及答案.doc
2023-2024学年福建省厦门市九年级上学期数学月考试题及答案.doc 2021-2022学年辽宁省沈阳市大东区九年级上学期语文期末试题及答案.doc
2021-2022学年辽宁省沈阳市大东区九年级上学期语文期末试题及答案.doc 2022-2023学年北京东城区初三第一学期物理期末试卷及答案.doc
2022-2023学年北京东城区初三第一学期物理期末试卷及答案.doc 2018上半年江西教师资格初中地理学科知识与教学能力真题及答案.doc
2018上半年江西教师资格初中地理学科知识与教学能力真题及答案.doc 2012年河北国家公务员申论考试真题及答案-省级.doc
2012年河北国家公务员申论考试真题及答案-省级.doc 2020-2021学年江苏省扬州市江都区邵樊片九年级上学期数学第一次质量检测试题及答案.doc
2020-2021学年江苏省扬州市江都区邵樊片九年级上学期数学第一次质量检测试题及答案.doc 2022下半年黑龙江教师资格证中学综合素质真题及答案.doc
2022下半年黑龙江教师资格证中学综合素质真题及答案.doc