中国科技论文在线
http://www.paper.edu.cn
MiRNA-BD:基于证据的 microRNA 生物标
志物识别模型#
林宇鑫1,2,吴文涛2,孙占东2,仇俊毅3,张文宇2,严文颖2,4,朱斐1,2,沈百荣2,4**
(1. 苏州大学计算机科学与技术学院,中国苏州,215006;
2. 苏州大学系统生物学研究中心,中国苏州,215006;
3. 苏州大学附属第一医院普外科,中国苏州,215006;
4. 太仓市正兴转化医学信息学中心,中国太仓,215400)
摘要:MicroRNA(miRNA)与生物体内许多重要的生物过程相关。大量研究表明,miRNA
可以作为疾病临床诊断和治疗的有效生物标志物。目前,miRNA 生物标志物的识别主要依
赖于生物学实验筛选以及生物信息学算法预测。这两类方法中,前者需要较大的时间和成
本开销,后者模型往往缺乏通用性。直到现在,还没有任何普适性的规则以用于 miRNA 生
物标志物的发掘。基于对 miRNA-mRNA 调控网络子结构的分析,发现了 miRNA 生物标志
物识别的两个重要证据并且构建了相应的通 用识别模型。与传统基于 miRNA 协同调控机制
的方法相比,该模型更加关注 miRNA 独立调控基因以及调控具有重要生物功能基因的能
力。目前,该模型已经在 MiRNA-BD 软件中实现并且成功应用至一些复杂疾病 miRNA 生
物标志物的识别中。
关键词:生物信息学;microRNA;生物标志物;模型构建
中图分类号:Q-31
MiRNA-BD: an evidence-based model for microRNA
biomarker discovery
LIN Yuxin1,2, WU Wentao2, SUN Zhandong2, QIU Junyi3, ZHANG Wenyu2, YAN
Wenying2,4, ZHU Fei1,2, SHEN Bairong2,4
(1. School of Computer Science & Technology, Soochow University, Suzhou, 215006, China;
2. Center for Systems Biology, Soochow University, Suzhou, 215006, China;
3. Department of General Surgery, The First Affiliated Hospital of Soochow University, Suzhou,
215006, China;
4. Taicang Center for Translational Bioinformatics, Taicang, 215400, China)
Abstract: MicroRNAs (miRNAs) are associated with many important biology processes and
plenty of studies have been reported on detecting miRNA as biomarkers for clinical diagnosis and
treatment. Most of the approaches are experimental, which are timing consuming and costly.
Bioinformatics models are therefore preferred and developed. However, until now, very few
models are evidence-based and no general rules can be applied to biomarker discovery. Based on
sub-structural analysis of miRNA-mRNA interaction network, two evidences are uncovered and
considered into a novel evidence-based model for miRNA biomarker discovery. Compared to
routine methods which focus on miRNA synergic functions, the model pays more attention to
miRNAs’ independent regulatory power and their biological functions. The model is implemented
in the program MiRNA-BD and its application in biomarker discovery for several diseases
demonstrated its predictive power.
Key words: Bioinformatics; MicroRNA; Biomarker; Model Construction
基金项目:国家自然科学基金(编号:31470821、31170795、91230117、81271378、81471488);高等学
校博士学科点专项科研基金资助课题(批准号:20113201110015)
作者简介:林宇鑫(1990-),男,硕士研究生,研究方向:疾病相关的统计模式识别及临床应用
通信联系人:沈百荣(1964-),男,教授,研究方向:医学系统生物学. E-mail: bairong.shen@suda.edu.cn
5
10
15
20
25
30
35
40
- 1 -
�
中国科技论文在线
0 引言
http://www.paper.edu.cn
45
50
55
60
65
MicroRNA(miRNA)是一类长度约 22-24 个核苷酸单位的非编码 RNA,它们能够在转
录后层次上调控基因表达。据报道,约有 60%的人类蛋白质编码基因受到 miRNA 调控,并
且很多疾病相关的生物过程也受到 miRNA 影响[1]。大量研究表明,miRNA 的异常表达与癌
症的发生发展密切相关,并且它们能够作为癌症临床诊断或预后的生物标志物[2-4]。
传统的 miRNA 生物标志物识别主要依赖于生物学实验。首先,从大规模高通量实验中
选择具有差异表达(Differentially expressed, DE)现象的 miRNA,然后通过低通量实验(例
如:实时定量 PCR 技术,即 real-time PCR)进一步验证得到最终的标志物[5, 6]。虽然实验分
析具有较高的准确性,但是由于样本资源有限、时间和成本开销较大等原因,这类方法仍然
具有一定的局限性。为此,一些生物信息学预测模型和算法被相继开发。例如,结合群组和
共调控之间的相关性分析,Madden 等人[7]提出了一个生物信息学模型鉴别疾病相关的
miRNA。Cun 和 FrÖhlich[8]设计并实现了一个 R 程序包,通过整合表达谱数据以及网络信息
发现潜在的生物标志物信号。仅管这些模型具有较高的研究价值,但是它们大多数基于“训
练-预测”的一般模式,预测结果极大地依赖于训练样本和训练数据。并且,在这些模型中
没有很好的普适性规则和机理能够被发掘和阐释。另一方面,疾病相关的 miRNA 并不完全
等价于生物标志物。因为生物标志物严格意义上属于健康到疾病状态改变的指示单元,它们
应当具有较高的灵敏度和较好的特异性[9]。
针对 miRNA 生物标志物的识别,很多工作都致力于 miRNA 的协同调控机制,很少有
研究关注于 miRNA-mRNA 调控网络的子结构,特别是网络中的“脆弱”结构。就像古希腊
神话故事中的英雄人物 Achilles heel 一样,他全身坚不可摧,唯有脚后跟是致命弱点。基于
之前的研究,我们发现 miRNA 具有一定的独立调控基因的能力,并且作为标志物的 miRNA
能够独立调控更多的基因[10]。在此基础上,结合具体的生物过程和生物功能,我们发现了
能够用于 miRNA 生物标志物识别的新的证据:作为生物标志物的 miRNA 能够调控更多的
转录因子(Transcription factor, TF)基因[11]。综合以上两点,我们构建并实现了基于证据的
生物信息学模型 MiRNA-BD,并将其应用至具体疾病 miRNA 生物标志物的识别中。
1 数据和方法
70
1.1 数据搜集
1.1.1 人类 miRNA 数据
75
人类 miRNA 数据全部从 miRBase 数据库(2014 年 06 月第 21 版)[12]中下载得到,包
括 2588 个成熟体和 1881 个前体。每个 miRNA 的 ID、miRBase 收录号、序列以及早期的命
名 ID 都涵盖其中。同时,我们使用 RNAfold 软件[13]预测得到所有前体的二级结构信息。
1.1.2 转录因子基因和 miRNA 生物标志物数据
转录因子基因来源于 Vaquerizas 等人[14]的研究,文章共收录了 1834 个转录因子基因。
同时,通过文献挖掘,我们收集了 180 个被明确报道作为癌症生物标志物的 miRNA,涵盖
了 20 多种不同的癌症类型。
1.2 人类 miRNA-mRNA 调控网络的重构
80
人类 miRNA-mRNA 调控网络中的 miRNA-mRNA 靶标对来源于实验验证和计算机预测
两类数据。为了保证数据的有效性和可靠性,每一类数据均选自目前被广泛认可的数据库。
- 2 -
�
中国科技论文在线
具体信息参见表 1。
http://www.paper.edu.cn
表 1 人类 miRNA-mRNA 靶标对数据来源
类型
实验验证
计算机预测
数据库名称
miRTarBase
TarBase
miRecords
miR2Disease
HOCTAR
ExprTargetDB
starBase
版本号
V4.5
V6.0
V4.0
—
V2.0
—
V2.0
引文
[15]
[16]
[17]
[18]
[19]
[20]
[21]
为了减少假阳性,实验验证的数据中我们主要选择被低通量实验验证的 miRNA-mRNA
靶标对;对于计算机预测得到的数据,我们选择那些在两个及以上预测数据库中同时存在的
miRNA-mRNA 靶标对用于后续的网络构建。同时,我们注意到 miRNA 的命名规则从
miRBase 第 17 版本开始被逐步改变。一些数据库(例如:miRTarBase,TarBase 和 starBase)
使用了新的规则命名 miRNA(例如:hsa-miR-93-5p,hsa-miR-93-3p),而仍有一部分(例
如:miRecords,miR2Disease,HOCTAR 和 ExprTargetDB)沿用了之前的命名规则(例如:
hsa-miR-93,hsa-miR-93*)。为此,我们构建了 miRNA 命名转换字典,并且在网络重构前
增加了数据“清洗”操作。最终,重构的网络涵盖了 48868 条 miRNA-mRNA 靶标对,其中
涉及到 618 个 miRNA 以及 9492 个靶基因。
2 结果
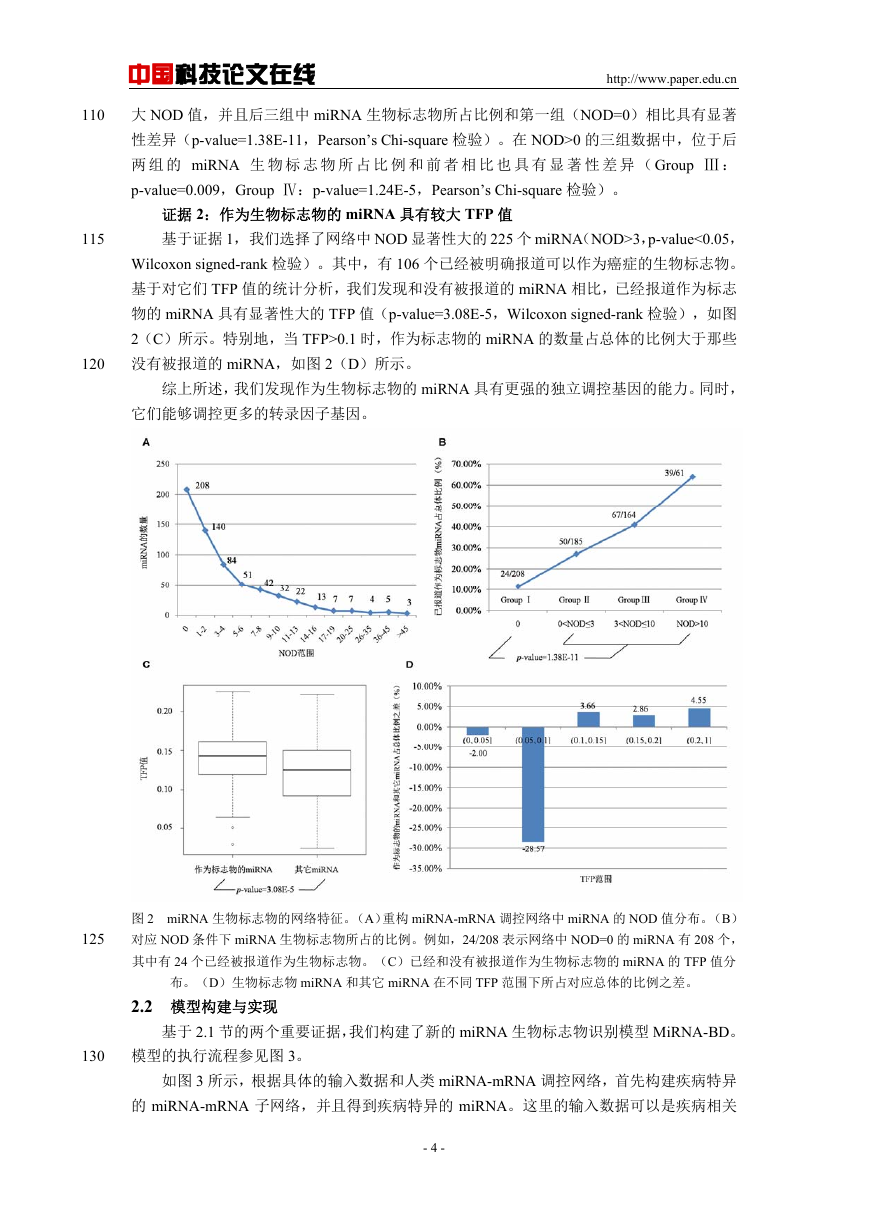
2.1 miRNA 生物标志物的网络特征
图 1 miRNA-mRNA 的调控模式。这里定义了四种类型:被独立/协同调控的转录因子基因;被独立/协同
调控的非转录因子基因。
如图 1 所示,基于网络的子结构,我们把 miRNA-mRNA 的调控模式分为四类。据此,
我们定义了两个评估标准以量化 miRNA 在网络中调控基因的能力:
标准 1:NOD(novel out degree),miRNA 独立调控基因的数量
标准 2:TFP(percentage of TF genes),miRNA 调控基因中转录因子基因所占的比例
以图 1 中四个 miRNA 为例,它们的 NOD 值分别为 1、0、1、2,TFP 值分别为 0、2/5、
1/2、1/2。在以上两个标准的基础上,通过计算重构网络中 618 个 miRNA 的 NOD 和 TFP
值,综合分析作为生物标志物的 miRNA 独立调控基因以及调控转录因子基因的情况,我们
发现了能够用于 miRNA 生物标志物识别的两个重要证据:
证据 1:作为生物标志物的 miRNA 具有较大 NOD 值
人类 miRNA-mRNA 调控网络中 618 个 miRNA 的 NOD 数值分布如图 2(A)所示。由
图可知,miRNA 的 NOD 数值在一定程度上也满足幂律分布。基于 NOD 值,我们将这些
miRNA 分为 4 组,如图 2(B)所示。统计结果表明,作为生物标志物的 miRNA 都具有较
- 3 -
85
90
95
100
105
�
中国科技论文在线
http://www.paper.edu.cn
110
115
120
大 NOD 值,并且后三组中 miRNA 生物标志物所占比例和第一组(NOD=0)相比具有显著
性差异(p-value=1.38E-11,Pearson’s Chi-square 检验)。在 NOD>0 的三组数据中,位于后
两 组 的 miRNA 生 物 标 志 物 所 占 比 例 和 前 者 相 比 也 具 有 显 著 性 差 异 ( Group Ⅲ :
p-value=0.009,Group Ⅳ:p-value=1.24E-5,Pearson’s Chi-square 检验)。
证据 2:作为生物标志物的 miRNA 具有较大 TFP 值
基于证据 1, 我们选择了网络中 NOD 显著性大的 225 个 miRNA(NOD>3,p-value<0.05,
Wilcoxon signed-rank 检验)。其中,有 106 个已经被明确报道可以作为癌症的生物标志物。
基于对它们 TFP 值的统计分析,我们发现和没有被报道的 miRNA 相比,已经报道作为标志
物的 miRNA 具有显著性大的 TFP 值(p-value=3.08E-5,Wilcoxon signed-rank 检验),如图
2(C)所示。特别地,当 TFP>0.1 时,作为标志物的 miRNA 的数量占总体的比例大于那些
没有被报道的 miRNA,如图 2(D)所示。
综上所述,我们发现作为生物标志物的 miRNA 具有更强的独立调控基因的能力。同时,
它们能够调控更多的转录因子基因。
图 2 miRNA 生物标志物的网络特征。(A)重构 miRNA-mRNA 调控网络中 miRNA 的 NOD 值分布。(B)
对应 NOD 条件下 miRNA 生物标志物所占的比例。例如,24/208 表示网络中 NOD=0 的 miRNA 有 208 个,
其中有 24 个已经被报道作为生物标志物。(C)已经和没有被报道作为生物标志物的 miRNA 的 TFP 值分
125
布。(D)生物标志物 miRNA 和其它 miRNA 在不同 TFP 范围下所占对应总体的比例之差。
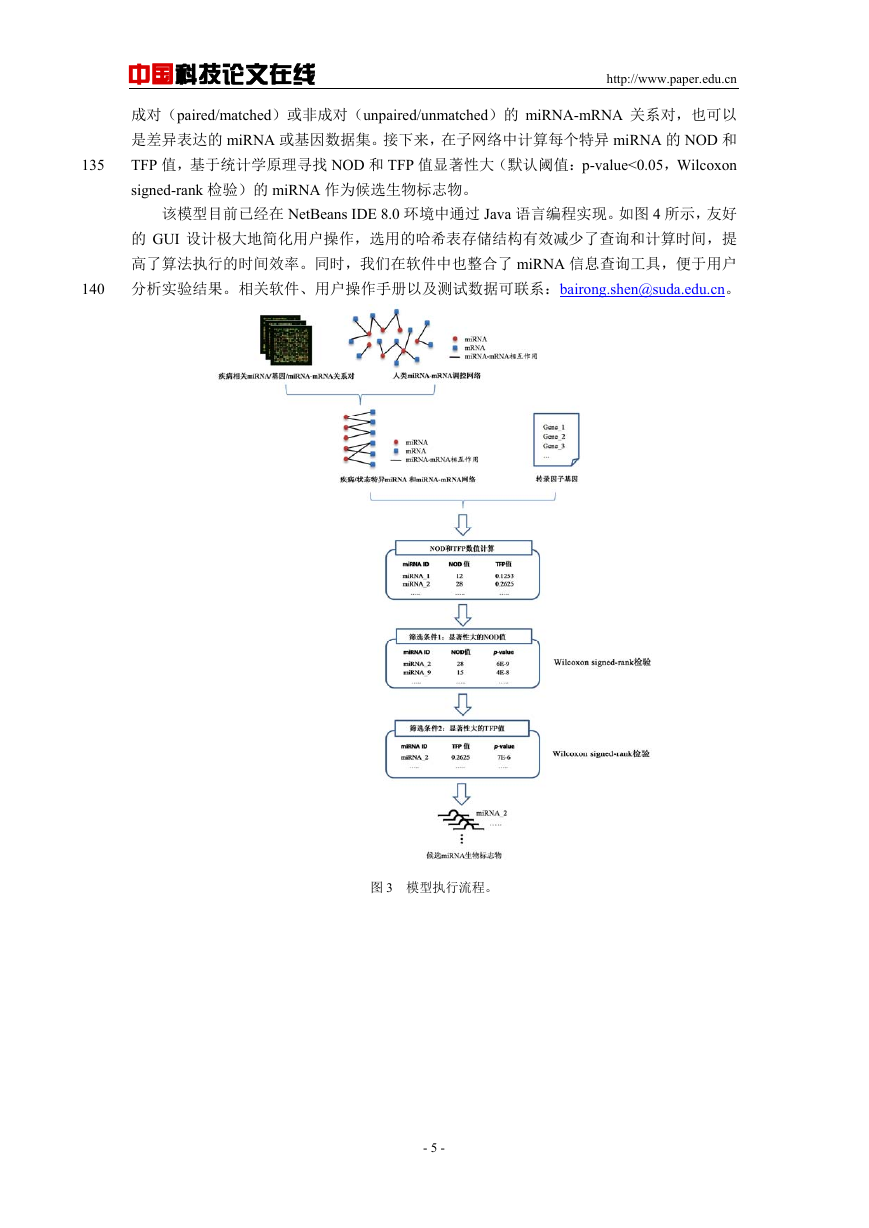
2.2 模型构建与实现
基于 2.1 节的两个重要证据,我们构建了新的 miRNA 生物标志物识别模型 MiRNA-BD。
130
模型的执行流程参见图 3。
如图 3 所示,根据具体的输入数据和人类 miRNA-mRNA 调控网络,首先构建疾病特异
的 miRNA-mRNA 子网络,并且得到疾病特异的 miRNA。这里的输入数据可以是疾病相关
- 4 -
�
中国科技论文在线
http://www.paper.edu.cn
成对(paired/matched)或非成对(unpaired/unmatched)的 miRNA-mRNA 关系对,也可以
是差异表达的 miRNA 或基因数据集。接下来,在子网络中计算每个特异 miRNA 的 NOD 和
TFP 值,基于统计学原理寻找 NOD 和 TFP 值显著性大(默认阈值:p-value<0.05,Wilcoxon
signed-rank 检验)的 miRNA 作为候选生物标志物。
该模型目前已经在 NetBeans IDE 8.0 环境中通过 Java 语言编程实现。如图 4 所示,友好
的 GUI 设计极大地简化用户操作,选用的哈希表存储结构有效减少了查询和计算时间,提
高了算法执行的时间效率。同时,我们在软件中也整合了 miRNA 信息查询工具,便于用户
分析实验结果。相关软件、用户操作手册以及测试数据可联系:bairong.shen@suda.edu.cn。
135
140
图 3 模型执行流程。
- 5 -
�
中国科技论文在线
http://www.paper.edu.cn
图 4 软件实现。
145
2.3 结果验证分析
之前的研究中,我们已经把相关模型应用至前列腺癌[10, 22]、胃癌[23]、儿童急性白血病[11]、
肾透明细胞癌[24]、脓毒症[25]等复杂疾病 microRNA 生物标志物的识别中,每种疾病我们选
用不少于两种方法验证实验结果的准确性和可靠性,相关信息参见表 2。
表 2 实验结果汇总
序号
疾病
样本
类型
标志物
数量
1
2
3
4
5
6
前列腺癌
前列腺癌
胃癌
儿童急性
白血病
肾透明
细胞癌
脓毒症
miRNA
mRNA
mRNA
miRNA
mRNA
miRNA
mRNA
miRNA
mRNA
miRNA
39
11
17
3
11
10
验证方法
文献
报道
q-PCR
实验
ROC
分析
富集
分析
√
√
√
√
√
√
√
√
√
√
√
√
√
√
√
√
√
引文
(PMID)
24618011
26540468
24982912
26317787
23841900
24809055
150
注:样本类型加粗项表示成对的(paired/matched)miRNA 和 mRNA 表达谱数据。
由表 2 可知,我们的模型适用于多种复杂疾病 miRNA 标志物的研究,通用性良好,并
且对于输入样本的类型没有限制。尽管成对的 miRNA 和 mRNA 数据更加严格,但是在非成
对的样本以及仅有 miRNA 或基因表达数据的情况下,模型也能得到较为准确的结果。
在之前的工作中,我们已将相关模型与当前较为主流的预测方法做了比较[10]。结果表
- 6 -
�
155
160
165
170
中国科技论文在线
http://www.paper.edu.cn
明,我们的方法优于 Kurahashi 等人提出的基于 t 检验的方法[26]。同时,相比较 Xu 等人提
出的基于 SVM 模型的方法[27],我们的模型不依赖于任何先验知识,具有相当的竞争性。
为了验证重构 miRNA-mRNA 网络的可靠性,我们仍然选择儿童急性白血病研究中使用
到的数据集[11],其中,miRNA 和 mRNA 数据分别来自 GSE35320[28]和 GSE43176[29]。我们
在 R 平台使用 Limma 包里的 eBayes 方法得到差异表达的 miRNA 和 mRNA(p-value<0.05),
基于重构的 miRNA-mRNA 网络构建疾病特异的 miRNA-mRNA 子网络,进而从子网络中筛
选出 NOD 和 TFP 都显著性大的 miRNA(p-value<0.05,Wilcoxon signed-rank 检验)。最终
的结果参见表 3。
表 3 基于重构 miRNA-mRNA 网络的预测结果
序号
1
2
3
miRNA ID
miR-155-5p
miR-196b-5p
miR-221-3p
NOD 值
NOD 的 p-value
37
28
17
9.69E-8
2.65E-5
0.016
TFP 值
0.1852
0.1733
0.1951
TFP 的 p-value
0.008
0.027
0.004
由表 3 可知,基于重构网络共得到了三个候选标志物:miR-155-5p、miR-196b-5p 和
miR-221-3p。其中, miR-155-5p 和 miR-196b-5p 在我们之前的研究中已经证实它们分别在
儿童白血病和 M4-M5 亚型中过表达[11]。PubMed 中检索发现,miR-221-3p 也与白血病密切
相关[30],并且它的初级体 pri-221 能够作为白血病的分子标志物[31]。综上所述,我们重构的
网络具有较好的鲁棒性。
考虑到某些疾病 miRNA 表达谱数据较难获取, Zhao 等人[32]提出了基于基因表达谱数
据反推筛选癌症相关 miRNA 的方法。为了进一步验证模型的灵活性和有效性,我们和他们
的方法做了比较。这里,我们选用了与其相同的数据集(参见表 4)筛选得到差异表达的基
因(p-value<0.05,t 检验)。
表 4 验证实验中使用到的数据集
癌症名称
GEO 收录号
样本数量(疾病组/对照组)
肺癌
结肠癌
胃癌
乳腺癌
GSE7670
GSE10072
GSE9348
GSE20916
GSE13911
GSE19826
GSE15852
GSE20437
54(27/27)
107(58/49)
82(70/12)
69(45/24)
69(38/31)
27(12/15)
86(43/43)
36(18/18)
实验平台
GPL96
GPL96
GPL570
GPL570
GPL570
GPL570
GPL96
GPL96
175
180
基于 MiRNA-BD 模型,每种癌症我们选择两组数据集预测结果中共同的 miRNA 作为
最终结果。同样,我们选择 HMDD 数据库(版本 2.0)[33]中的数据作为预测精度的评价标
准,并基于此在 PubMed 中进一步检索 miRNA 是否与对应癌症相关(检索时间:2015 年 11
月 15 日),如图 5 所示。
由图可知,针对上述四种癌症,我们方法的预测精度和 Zhao 等人的方法相近。值得注
意的,Zhao 等人的方法基于基因表达谱数据和信号转导通路聚类,同时,最终 HRmiRs 结
果中的 miRNA 是两组数据集中 Ms(miRNA score)较大部分的交集,并非两组数据结果的
直接交集。而我们的方法基于统计证据,不依赖任何先验知识,计算时间短,相对简单高效。
更为重要的,当存在研究疾病缺少基因表达谱数据或表达谱数据质量不高(例如:样本量过
少、样本数据背景噪声较大等)等情况时,我们的方法能够基于 miRNA 表达谱数据精准预
- 7 -
�
中国科技论文在线
http://www.paper.edu.cn
测,具有显著优势。综上所述,我们的模型具有较好的灵活性和预测的准确性。
185
190
图 5 预测精度比较。(A)基于 HMDD 数据库检索;(B)基于 PubMed 数据库检索。
3 讨论
我们发现了两个可以用于 miRNA 生物标志物识别的重要证据。其一,基于网络结构分
析,明显地,miRNA 对基因的单独调控成分属于 miRNA-mRNA 网络中的“脆弱”结构,
同时它对于系统状态的改变具有较大影响。其二,我们基于生物功能分析,转录因子基因是
基因网络中的关键因子,越多的转录因子参与调控,则对生物系统基因表达的影响就越显著。
综合实验分析,结果表明我们的证据和模型能够很好地适用于复杂疾病 miRNA 生物标
志物的识别,具有一定的通用性和可靠性。
195
参考文献
[1] Esteller, M., Non-coding RNAs in human disease[J]. Nat Rev Genet, 2011. 12(12): 861-74.
[2] Deng, Z.Q., et al., Expression level of miR-93 in formalin-fixed paraffin-embedded tissues of breast cancer
patients[J]. Genet Test Mol Biomarkers, 2014. 18(5): 366-70.
[3] Oue, N., et al., High miR-21 expression from FFPE tissues is associated with poor survival and response to
adjuvant chemotherapy in colon cancer[J]. Int J Cancer, 2014. 134(8): 1926-34.
[4] Diaz-Beya, M., et al., MicroRNA expression at diagnosis adds relevant prognostic information to molecular
categorization in patients with intermediate-risk cytogenetic acute myeloid leukemia[J]. Leukemia, 2014. 28(4):
804-12.
[5] Kojima, S., et al., MiRNA profiling in prostate cancer[J]. Nihon Rinsho, 2011. 69 Suppl 5: 92-5.
[6] Rahmann, S., et al., Identifying transcriptional miRNA biomarkers by integrating high-throughput sequencing
and real-time PCR data[J]. Methods, 2013. 59(1): 154-63.
[7] Madden, S.F., et al., Detecting microRNA activity from gene expression data[J]. BMC Bioinformatics, 2010.
11: 257.
[8] Cun, Y. and H. Frohlich, netClass: an R-package for network based, integrative biomarker signature
discovery[J]. Bioinformatics, 2014. 30(9): 1325-6.
[9] Chen, J., M. Sun, and B. Shen, Deciphering oncogenic drivers: from single genes to integrated pathways[J].
200
205
210
- 8 -
�














 2023年江西萍乡中考道德与法治真题及答案.doc
2023年江西萍乡中考道德与法治真题及答案.doc 2012年重庆南川中考生物真题及答案.doc
2012年重庆南川中考生物真题及答案.doc 2013年江西师范大学地理学综合及文艺理论基础考研真题.doc
2013年江西师范大学地理学综合及文艺理论基础考研真题.doc 2020年四川甘孜小升初语文真题及答案I卷.doc
2020年四川甘孜小升初语文真题及答案I卷.doc 2020年注册岩土工程师专业基础考试真题及答案.doc
2020年注册岩土工程师专业基础考试真题及答案.doc 2023-2024学年福建省厦门市九年级上学期数学月考试题及答案.doc
2023-2024学年福建省厦门市九年级上学期数学月考试题及答案.doc 2021-2022学年辽宁省沈阳市大东区九年级上学期语文期末试题及答案.doc
2021-2022学年辽宁省沈阳市大东区九年级上学期语文期末试题及答案.doc 2022-2023学年北京东城区初三第一学期物理期末试卷及答案.doc
2022-2023学年北京东城区初三第一学期物理期末试卷及答案.doc 2018上半年江西教师资格初中地理学科知识与教学能力真题及答案.doc
2018上半年江西教师资格初中地理学科知识与教学能力真题及答案.doc 2012年河北国家公务员申论考试真题及答案-省级.doc
2012年河北国家公务员申论考试真题及答案-省级.doc 2020-2021学年江苏省扬州市江都区邵樊片九年级上学期数学第一次质量检测试题及答案.doc
2020-2021学年江苏省扬州市江都区邵樊片九年级上学期数学第一次质量检测试题及答案.doc 2022下半年黑龙江教师资格证中学综合素质真题及答案.doc
2022下半年黑龙江教师资格证中学综合素质真题及答案.doc