DTI
1 数据转换包括 3DT1 和 DTI 的转化(124)
a.在 Mriconvert 中将 3DT1 转换成 nifti 格式(2)注意原格式中必须全为横断位
图像。
b. 在 Mriconvert 中 将 DTI 数 据 首 先 转 换 FSL nifti 格 式 得 到 梯 度 信 息
_vbesc,(248+1)删除其他文件只留下_vbesc 文件,再继续将 DTI 格式转换成
nifti 格式以便用于追踪。(248+1)
c.在 Matlab 中查看梯度信息:load
name..vbesc
data=name..vbesc(:,1:4:end)',%%每隔 4 列是一样的所以只
取一列
2 DTI 头动校正,将校正后的 DTI 弥散图像转换成 4D 文件
outputfomat:FSL(4Dniftinii)->file:nifti3D->4D
(4Dname2DTI.nii1+r.*248+dti_ep.*248+1.vbesc+1.rp.txt)
3 结 构 像 配 准 事 3DT1 像 的 维 度 和 b0 像 的 维 度 一 样 。
Coregister(estimate&reslice)-> Reference: 选 择 头 动 矫 正 后 的 B0 像 ->
Images to Reslice: 选择 MRIConvert 转换的 3D T1 像. (2+r.*2)
4.结构像分割 data:选择配准后的结构像。(12=产生灰质和白质文件 c1* 2 和
c2* 2+seg_inv_sn.mat1+seg_sn.mat1+mr.*2+4)
二 纤维素追踪
1 由于纤维束追踪是在 DTI native space 进行的,所以有必要将标准空间的 ROI
投回到 DTI native space,以便达到空间位置对齐
a. Normalize(estimate &write)-> Data->Subject-> Source Image:矫正后的
3D T1 像-> Images to Write:矫正后的 3D T1 像。->Template Image:T1 模板。
结果产生 15=Normalize 参数*_sn.mat+ wmr.*2+12
b. 选择 deformation, 点击 Current Item 下 Inverse,然后选择 Current Module
下 Composition->Inverse->Composition , 再 次 选 择 Current Item 下 New :
Imported _sn.mat。Parameter File:矫正后 3D T1 标准化产生的_sn.mat 参数。
Image to base inverse on:矫正后的 3D T1 像。Apply to:需要空间变换的
ROI(aal)。 Interpolation:Nearest Neighbour。(w)
�
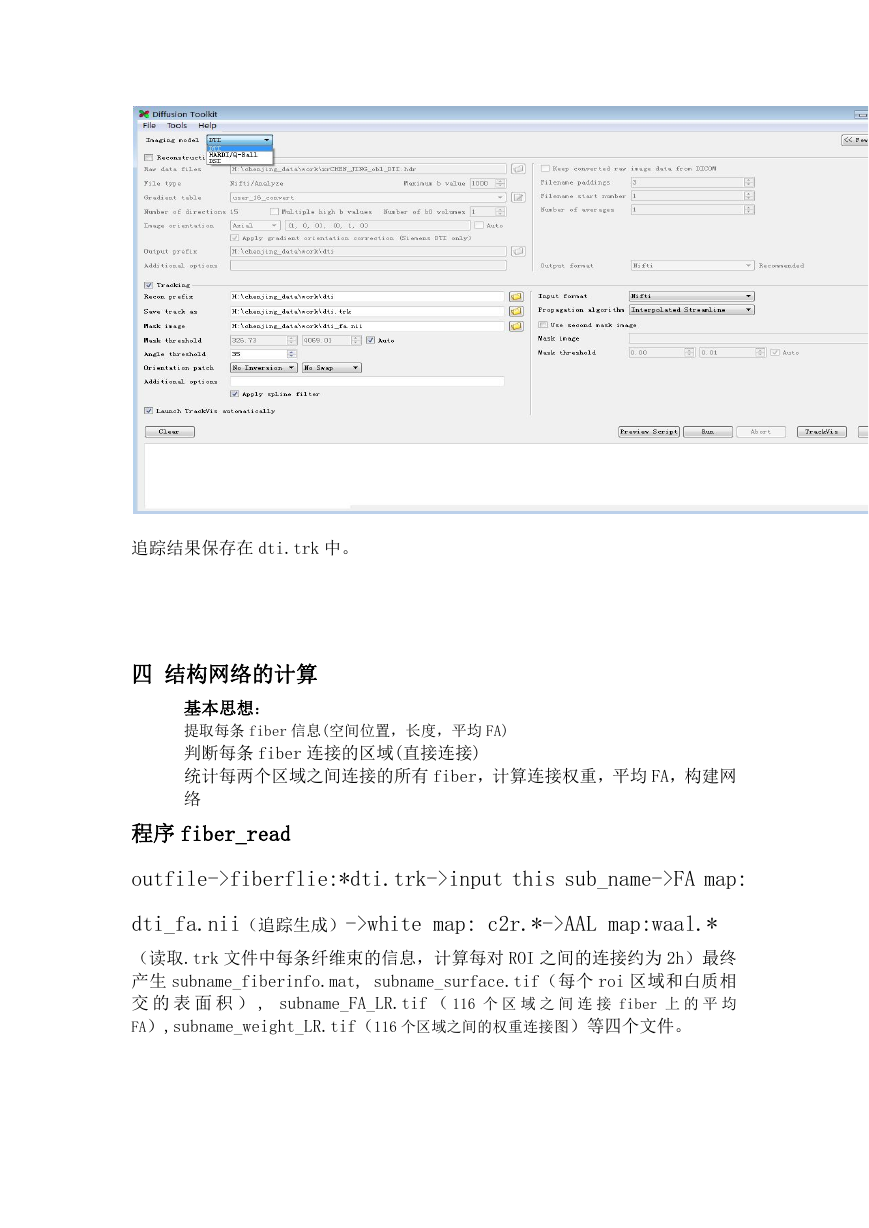
三 DTI 追踪(Toolkit&trackvis)
纤维束追踪主要是在软件 Toolkit&trackvis 下完成的,该软件包括追踪和显示
两个部分,Toolkit 用于追踪,Trackvis 用于纤维显示和统计,这里我们主要利
用其追踪结果来构建结构网络。
1
name.bvesc->data=name(:,1:4:end)'->dlmwrite('name.txt',data),
每隔 4 行相同的情况下只需取其中一行并转置,提取梯度信息。
load
%%假设
,
利
用
指
令
�
追踪结果保存在 dti.trk 中。
四 结构网络的计算
基本思想:
提取每条 fiber 信息(空间位置,长度,平均 FA)
判断每条 fiber 连接的区域(直接连接)
统计每两个区域之间连接的所有 fiber,计算连接权重,平均 FA,构建网
络
程序 fiber_read
outfile->fiberflie:*dti.trk->input this sub_name->FA map:
dti_fa.nii(追踪生成)->white map: c2r.*->AAL map:waal.*
(读取.trk 文件中每条纤维束的信息,计算每对 ROI 之间的连接约为 2h)最终
产生 subname_fiberinfo.mat, subname_surface.tif(每个 roi 区域和白质相
交 的 表 面 积 ) , subname_FA_LR.tif ( 116 个 区 域 之 间 连 接 fiber 上 的 平 均
FA),subname_weight_LR.tif(116 个区域之间的权重连接图)等四个文件。
�





 2023年江西萍乡中考道德与法治真题及答案.doc
2023年江西萍乡中考道德与法治真题及答案.doc 2012年重庆南川中考生物真题及答案.doc
2012年重庆南川中考生物真题及答案.doc 2013年江西师范大学地理学综合及文艺理论基础考研真题.doc
2013年江西师范大学地理学综合及文艺理论基础考研真题.doc 2020年四川甘孜小升初语文真题及答案I卷.doc
2020年四川甘孜小升初语文真题及答案I卷.doc 2020年注册岩土工程师专业基础考试真题及答案.doc
2020年注册岩土工程师专业基础考试真题及答案.doc 2023-2024学年福建省厦门市九年级上学期数学月考试题及答案.doc
2023-2024学年福建省厦门市九年级上学期数学月考试题及答案.doc 2021-2022学年辽宁省沈阳市大东区九年级上学期语文期末试题及答案.doc
2021-2022学年辽宁省沈阳市大东区九年级上学期语文期末试题及答案.doc 2022-2023学年北京东城区初三第一学期物理期末试卷及答案.doc
2022-2023学年北京东城区初三第一学期物理期末试卷及答案.doc 2018上半年江西教师资格初中地理学科知识与教学能力真题及答案.doc
2018上半年江西教师资格初中地理学科知识与教学能力真题及答案.doc 2012年河北国家公务员申论考试真题及答案-省级.doc
2012年河北国家公务员申论考试真题及答案-省级.doc 2020-2021学年江苏省扬州市江都区邵樊片九年级上学期数学第一次质量检测试题及答案.doc
2020-2021学年江苏省扬州市江都区邵樊片九年级上学期数学第一次质量检测试题及答案.doc 2022下半年黑龙江教师资格证中学综合素质真题及答案.doc
2022下半年黑龙江教师资格证中学综合素质真题及答案.doc