KEGG 数据库的使用方法与介绍 http://www.genome.jp/
KEGG 的数据
KEGG 中的 pathway 是根据相关知识手绘的,这里的手绘的意思可能是指人
工以特定的语言格式来确定通路各组件的联系;基因组信息主要是从 NCBI 等数
据库中得到的,除了有完整的基因序列外,还有没完成的草图;另外 KEGG 中
有一个“专有名词”KO(KEGG Orthology),它是蛋白质(酶)的一个分类体
系,序列高度相似,并且在同一条通路上有相似功能的蛋白质被归为一组,然后
打上 KO(或 K)标签。下面就首先来讲一下 KEGG orthology。
任找一个代谢通路图,在上方有 pathway meue | payhway entry
| Show(Hide) description | 这 3 个选项,点击 pathway entry, 出现了一个页面,这
个随时被连接出来的页面相信大家一定再熟悉不过了。在这个页面中的 pathway
map 项中点击按钮状的链接 Ortholog table 。就进入了 Ortholog table 如下的页面:
在这个表中,行与物种对应,3 个字母都是相应物中的英文
单词缩写,比如 has 表示 Homo sapiens,mcc 表示 Macaca mulatta;列就表示相
应的 Ortholog 分类,比如 K00844 就表示生物体内的己糖激酶 hexokinase 这一
类序列和功能相似的蛋白质类(酶类)。如上图 has 后有 3101,3098,3099 这 3
个条目,它表示在人类细胞中中存在 3 中不同的己糖激酶,它们分别由以上这 3
组数字代表的基因所编码,这 3 组数字应该是这 3 个基因的登录号。空白则表示
在该物种中不存在这种酶。
�
点击 K00844 则这一 KO 分类信息及成员列表都可显示出来;
点击 has 则链接到物种(人类)基因组去了;点击 P,则显示相应的代谢通路。
下面我们点击 3101,如下:
如上图,就是我们常见的一个页面,3101 是 KEGG 中的基因
ID(登录号), H.sapiens 表示物种,然后是基因的名称,表达的酶,属于哪
个 KO 分类以及参与哪些代谢途径;下面还有结构、序列信息等等。
所以从 Ortholog table 中可以很容易地知道一张代谢通路
上有哪些 KO 分类(酶类),并且这些酶类的成员在各物种中分配存在的情况以
及特定的名称。
怎么看 KEGG 中代谢通路图
�
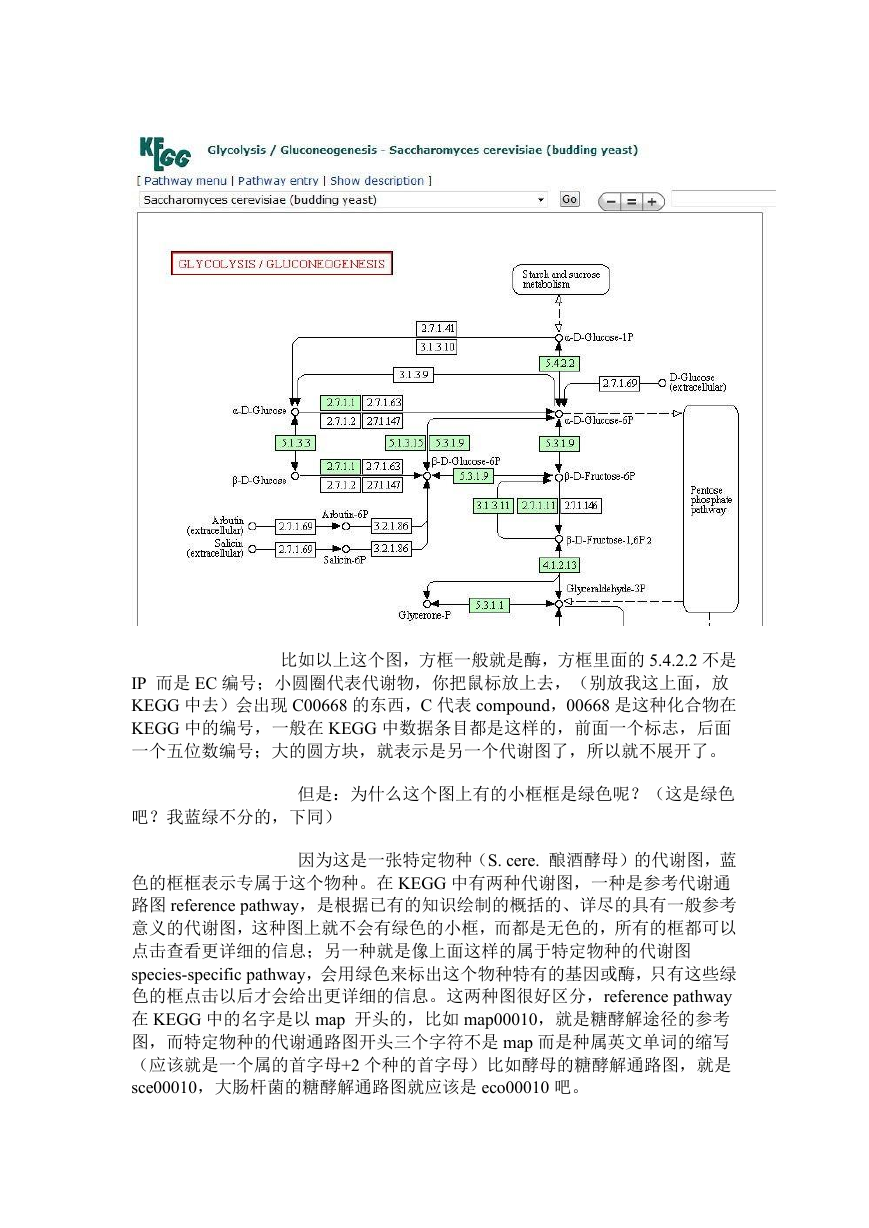
比如以上这个图,方框一般就是酶,方框里面的 5.4.2.2 不是
IP 而是 EC 编号;小圆圈代表代谢物,你把鼠标放上去,(别放我这上面,放
KEGG 中去)会出现 C00668 的东西,C 代表 compound,00668 是这种化合物在
KEGG 中的编号,一般在 KEGG 中数据条目都是这样的,前面一个标志,后面
一个五位数编号;大的圆方块,就表示是另一个代谢图了,所以就不展开了。
吧?我蓝绿不分的,下同)
但是:为什么这个图上有的小框框是绿色呢?(这是绿色
因为这是一张特定物种(S. cere. 酿酒酵母)的代谢图,蓝
色的框框表示专属于这个物种。在 KEGG 中有两种代谢图,一种是参考代谢通
路图 reference pathway,是根据已有的知识绘制的概括的、详尽的具有一般参考
意义的代谢图,这种图上就不会有绿色的小框,而都是无色的,所有的框都可以
点击查看更详细的信息;另一种就是像上面这样的属于特定物种的代谢图
species-specific pathway,会用绿色来标出这个物种特有的基因或酶,只有这些绿
色的框点击以后才会给出更详细的信息。这两种图很好区分,reference pathway
在 KEGG 中的名字是以 map 开头的,比如 map00010,就是糖酵解途径的参考
图,而特定物种的代谢通路图开头三个字符不是 map 而是种属英文单词的缩写
(应该就是一个属的首字母+2 个种的首字母)比如酵母的糖酵解通路图,就是
sce00010,大肠杆菌的糖酵解通路图就应该是 eco00010 吧。
�
那么:怎么找这两种图呢?
(1)有下拉列表的时候,在列表选择 reference 或者是特定物种即可。
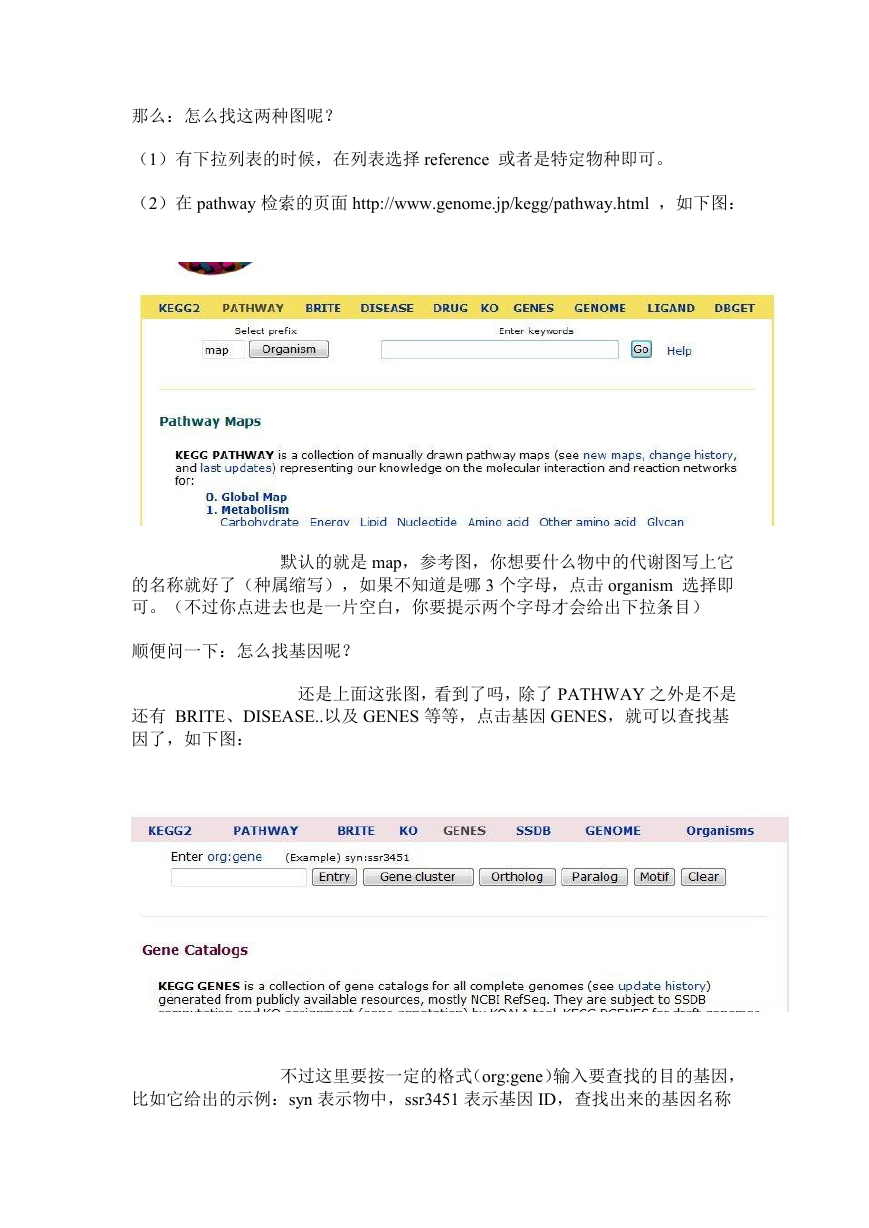
(2)在 pathway 检索的页面 http://www.genome.jp/kegg/pathway.html ,如下图:
默认的就是 map,参考图,你想要什么物中的代谢图写上它
的名称就好了(种属缩写),如果不知道是哪 3 个字母,点击 organism 选择即
可。(不过你点进去也是一片空白,你要提示两个字母才会给出下拉条目)
顺便问一下:怎么找基因呢?
还是上面这张图,看到了吗,除了 PATHWAY 之外是不是
还有 BRITE、DISEASE..以及 GENES 等等,点击基因 GENES,就可以查找基
因了,如下图:
比如它给出的示例:syn 表示物中,ssr3451 表示基因 ID,查找出来的基因名称
不过这里要按一定的格式(org:gene)输入要查找的目的基因,
�
是 psbE。其实我试了一下,若直接检索基因名称(而不是 KEGG 中的基因 ID)
syn:psbE 也是一样的。因为我不知道 KEGG 中基因 ID 如何编制的,但是,我同
时也不知道基因的名称是如何定义的。比如果糖 1,6-二磷酸酶 Fructose
1,6-biphosphatase 的基因就叫 fbp,我放进去能检索,但是我把有名的 gal 填上去
就不能检索,当然这可能与基因后面的乱七八糟的序号后缀有关,比如填上 gal1
就能检索了,所以我真不知道基因到底怎么命名的?当然我在 syn 中没找到 gal1
在 sce 中检索到了,这也说明了基因果然不是乱长的。
依旧是上面这个图,看到 KEGG2 了吗?点击。也会出现检
索框,这是一个总体性地检索框,在这里面输入关键词,代谢通路也好,glycolysis
也好, gal 也好,化合物也好,没那么多限制,KEGG 中的相关东西都会检索出
来,在这里浏览一下,再进行后续检索,也是一个不错的方法。
当然,代谢通路图,还有其他的查看形式(比如以 KO 查看),
以及图上可以点击,链接到这链接到那,点来点去总能点出奇怪的页面来,熟悉
一下也就熟悉了,这些东西会很有用,所以我就不说了。下面讲一下 KEGG 的
自动注释功能。
KEGG 的自动注释
KEGG Automatic Annotation Server,KEGG 的自动注释服务简称 KAAS。
在线网址为 http://www.genome.jp/tools/kaas/ 。就是你提交一段蛋白质序列或者
基因序列(必须是 fasta 格式),它自动在内部进行相似性比对,找到最相似的
基因,并确定检索基因的 KO 分类,然后给出这些基因所在的代谢通路并以以不
同的颜色标示这些基因。如下图:
�
我在 help 中随便复制了它的两条示例氨基酸序列,然后粘贴
到检索框中,进行了检索。检索框默认的蛋白质序列,如果不是的话要改选。然
后填上一个邮箱地址,点击又下角的 compute 即可。不出意外的话,你在接下来
的页面中应该看不到任何结果,甚至连提示都没有,原来它把结果发到你邮箱去
了。我也不明白就一个网页链接为什么还硬要发送到邮箱。 首先发你一封信说
已经接受,并给你一个期待结果显示的网址,一段时间后,会发你另外一封邮件,
说已经完成。打开它给的网址,就能看到结果了,如下:
�
看来从 1:20 开始计算到 1:50 才结束,两条氨基酸链计算
了 30 分钟(不过我感觉没这么长呀)。人家说了,计算时间是与要和检索序列
对比的目标序列成正比,因此在检索的时候最好限制一下检索范围。
点击 html 有两条代谢通量图的条目,点开他们就可以直观
地看出我们检索的未知序列在代谢通路中的位置和作用了。Text 给出的是两个
KO 分类。
好像北京大学的生命科学学院也搞了一个 KOBA,也是基于 KEGG 中的 KO 进
行注释的一个服务,应该和这个差不多吧。
代谢通路的着色
或者基因(酶)着色以高亮显示呢?
怎么在 KEGG 检索出来的代谢通路中给特定的一些化合物
进入网页
http://www.genome.jp/kegg/tool/color_pathway.html ,或者由 pathway 主页的 Color
objects in KEGG pathways 进入,看图:
�
如上图,search against 下拉出你可供选择的代谢通量图,总
所周知的一个很烦人的问题就是,在这些下拉列表中,条目排序竟然是乱七八糟
的很难索引。还好我发现把焦点定在这个下拉列表的最顶端的文本框上(即文本
框变成选中的蓝色),然后在键盘上拼写你要的那个物中的英文单词,只需要拼
两三个字符相应的代谢通量图就出现在顶端了。比如我要找酵母的代谢通量图,
只需要在文本框变蓝的时候拼写“sacc”这几个字符“Saccharomyces
cerevisiae(budding yeast)”就自动被置于上面了。或者不把焦点集中在文本框中
也行,但是你要很快地拼写 sacc,否者的话焦点会在以这几个字符开头的条目之
间切换。
如上图,右边有示例,这个貌似不要太简单。想给谁着色就
把它写出来后面跟上颜色就好了,一个一行。比如写上 C00118 blue 就表示在代
谢通路图中把 C00118 这种代谢物(3-磷酸甘油醛,GAP)给着上蓝色。但是大
家也看出来了,着色可以自定义背景色,也可以同时定义前景色。我曾一度琢磨
前景色是干嘛的,琢磨半天发现没用。背景色就是把方框或者圆圈涂成选定的颜
色,这自然是要的;而前景色是谁的颜色,就是方框里面的 5.4.2.2 这几个数字
的颜色,或者是小圆圈圆周的颜色,这有必要定义吗,所以后面直接跟一种颜色
就行了。
然后就可以了。我随便弄个 gal1 想去着色,KEGG 突然
说在酵母中找不到 gal1,怎么可能找不到呢?我前面还在 GENES 中搜过呢,分
明是酵母,分明是 gal1,分明搜的到,我当时还大为兴叹,唉,看来基因果然不
�













 2023年江西萍乡中考道德与法治真题及答案.doc
2023年江西萍乡中考道德与法治真题及答案.doc 2012年重庆南川中考生物真题及答案.doc
2012年重庆南川中考生物真题及答案.doc 2013年江西师范大学地理学综合及文艺理论基础考研真题.doc
2013年江西师范大学地理学综合及文艺理论基础考研真题.doc 2020年四川甘孜小升初语文真题及答案I卷.doc
2020年四川甘孜小升初语文真题及答案I卷.doc 2020年注册岩土工程师专业基础考试真题及答案.doc
2020年注册岩土工程师专业基础考试真题及答案.doc 2023-2024学年福建省厦门市九年级上学期数学月考试题及答案.doc
2023-2024学年福建省厦门市九年级上学期数学月考试题及答案.doc 2021-2022学年辽宁省沈阳市大东区九年级上学期语文期末试题及答案.doc
2021-2022学年辽宁省沈阳市大东区九年级上学期语文期末试题及答案.doc 2022-2023学年北京东城区初三第一学期物理期末试卷及答案.doc
2022-2023学年北京东城区初三第一学期物理期末试卷及答案.doc 2018上半年江西教师资格初中地理学科知识与教学能力真题及答案.doc
2018上半年江西教师资格初中地理学科知识与教学能力真题及答案.doc 2012年河北国家公务员申论考试真题及答案-省级.doc
2012年河北国家公务员申论考试真题及答案-省级.doc 2020-2021学年江苏省扬州市江都区邵樊片九年级上学期数学第一次质量检测试题及答案.doc
2020-2021学年江苏省扬州市江都区邵樊片九年级上学期数学第一次质量检测试题及答案.doc 2022下半年黑龙江教师资格证中学综合素质真题及答案.doc
2022下半年黑龙江教师资格证中学综合素质真题及答案.doc