MEGA 软件构建系统发育树
摘要 :以白色念珠菌属下面的十个种的 18s RNA 为例,构建系统发育树来说明 MEGA
软件的使用方法。
1 背景简介
1.1 MEGA(分子进化遗传分析)
MEGA 的全称是 Molecular Evolutionary Genetics Analysis。MEGA is an
integrated tool for automatic and manual sequence alignment, inferring
phylogenetic trees, mining web-based databases, estimating rates of molecular
evolution, and testing evolutionary hypotheses. MEGA 可用于序列比对、进化树
的推断、估计分子进化速度、验证进化假说等。MEGA 还可以通过网络(NCBI)进行序
列的比对和数据的搜索。
最新版本:MEGA 5.1 Beta (软件开发者建议其结果不用于发表文章)
建议下载版本:MEGA 5.05 for Windows and Mac OS。
MEGA 5 has been tested on the following Microsoft Windows® operating systems:
Windows 95/98, NT, 2000, XP, Vista, version 7, Linux and Mac OS [1].
MEGA 5.05 可免费下载,只需输入名字及有效邮箱,下载链接会发送至邮箱,点击
可下载。
1.2 系统发育树定义
系统发育树(英文:Phylogenetic tree)又称为演化树(evolutionary tree),是表明被
认为具有共同祖先的各物种间演化关系的树。是一种亲缘分支分类方法(cladogram)。
在树中,每个节点代表其各分支的最近共同祖先,而节点间的线段长度对应演化距离(如
估计的演化时间)
1.3 系统发育树的分类
根据有根和无根来区分:树可分为有根树和无根树两类。有根树是具有方向的
树, 根据系统发生树可推断出物种的起源包含唯一的节点,将其作为树中所有物
种的最近共同祖先。最常用的确定树根的方法是使用一个或多个无可争议的同源物
种作为外群(英文 outgroup),这个外群要足够近,以提供足够的信息,但又不
�
能太近以至于和树中的种类相混。把有根树去掉根即成为无根树。一棵无根树在没
有其他信息(外群)或假设(如假设最大枝长为根)时不能确定其树根。无根树是
没有方向的,其中线段的两个演化方向都有可能。
基于单个同源基因差异构建的系统发生数应称之为基因树。因为这种树代表的
仅仅是单个基因的进化历史。而不是它所在物种的进化历史。物种树一般最好是从
多个基因数据的分析中得到。例如一项关于植物进化的研究中,用了 100 个不同的
基因来构建物种树,因为进化是发生在生物体种群水平上的,而不是发生在个体水
平上的,虽然表面上不需要更多的数据,但实际上还是有必要的。基因树和物种树
之间的差异是很重要的,如果只用等位基因来构建物种数,那许多人人和大猩猩就
会分到一起,而不是和其他人分到一起。
1.4 构建方法
要构建一个进化树(phyligenetic tree)。构建进化树的算法主 要分为两类:
独立元素法(discrete character methods)和距离依靠法(distance methods)。
所谓独立元素法是指进化树的拓扑形状是由序列上的每个状态决定的,而距离依靠
法是指进化树的拓扑形状由两两序列的进化距离决定的。进化树枝条的长度代表着
进化距离。独立元素法包括最大简约性法(Maximum Parsimony methods)和最大
可能性法(Maximum Likelihood methods);距离依靠法包括除权配对法(UPGMAM)
和邻位相连法(Neighbor-joining)。
2 蛋白质序列分析使用方法
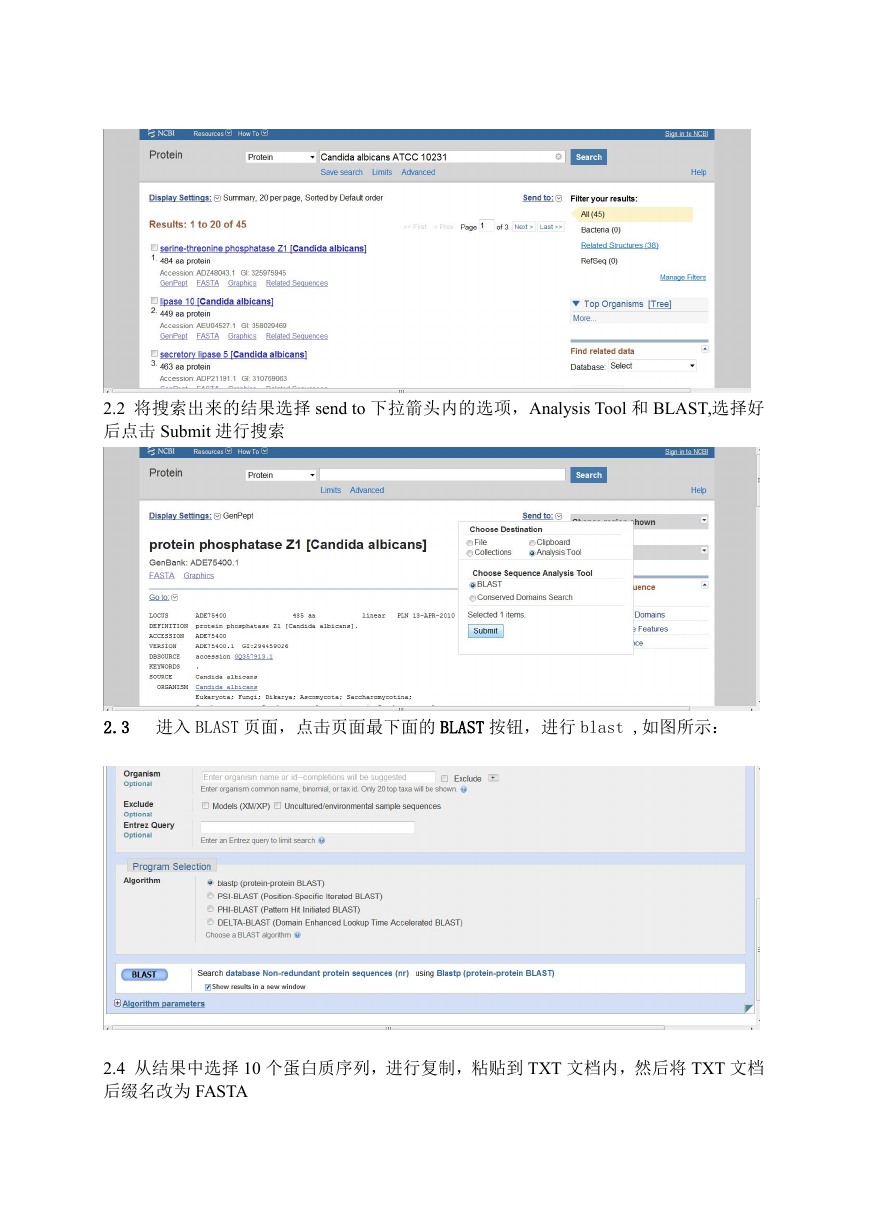
2.1 打开网址 http://www.ncbi.nlm.nih.gov/protein/,将菌名输入到 protein 后面的框内,
点 Search 键,选择一个搜索结果点击进入
�
2.2 将搜索出来的结果选择 send to 下拉箭头内的选项,Analysis Tool 和 BLAST,选择好
后点击 Submit 进行搜索
2.3 进入 BLAST 页面,点击页面最下面的 BLAST 按钮,进行 blast ,如图所示:
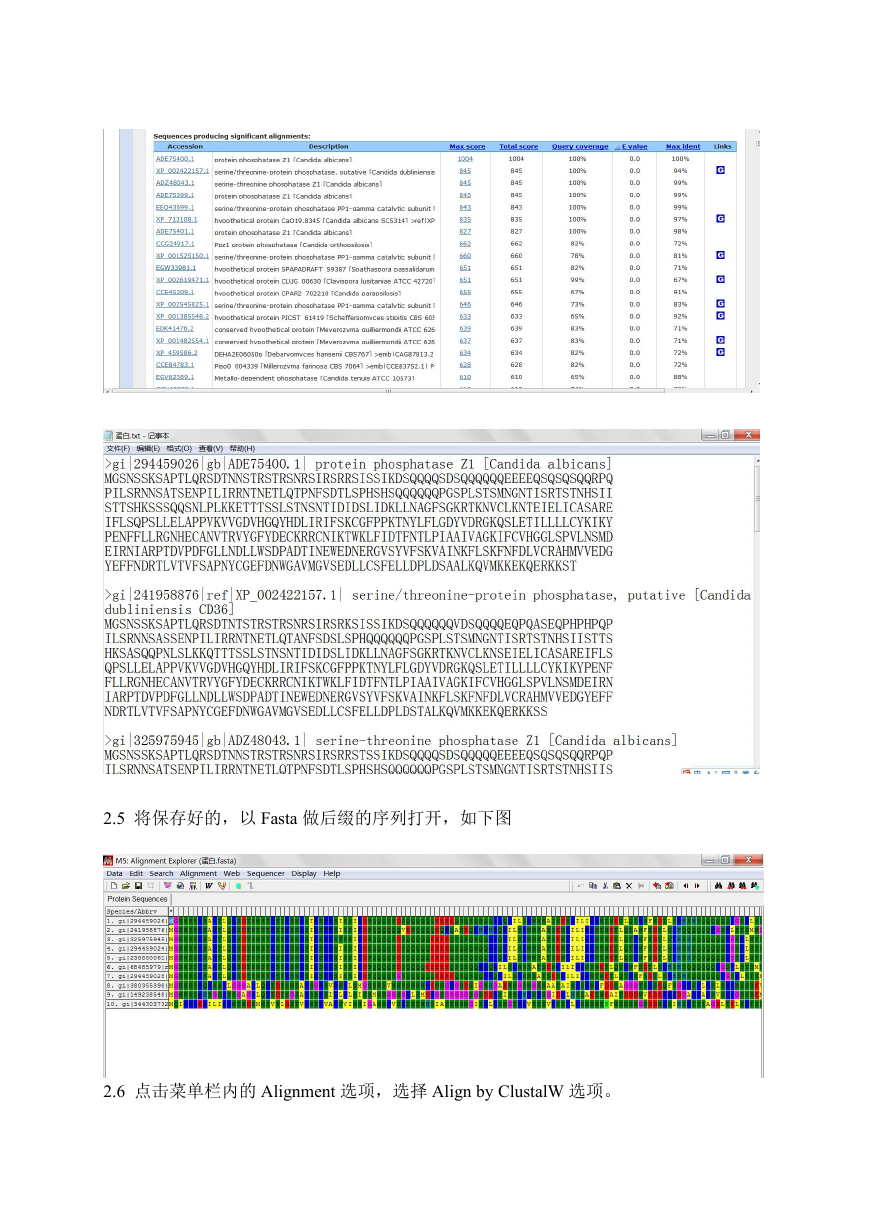
2.4 从结果中选择 10 个蛋白质序列,进行复制,粘贴到 TXT 文档内,然后将 TXT 文档
后缀名改为 FASTA
�
2.5 将保存好的,以 Fasta 做后缀的序列打开,如下图
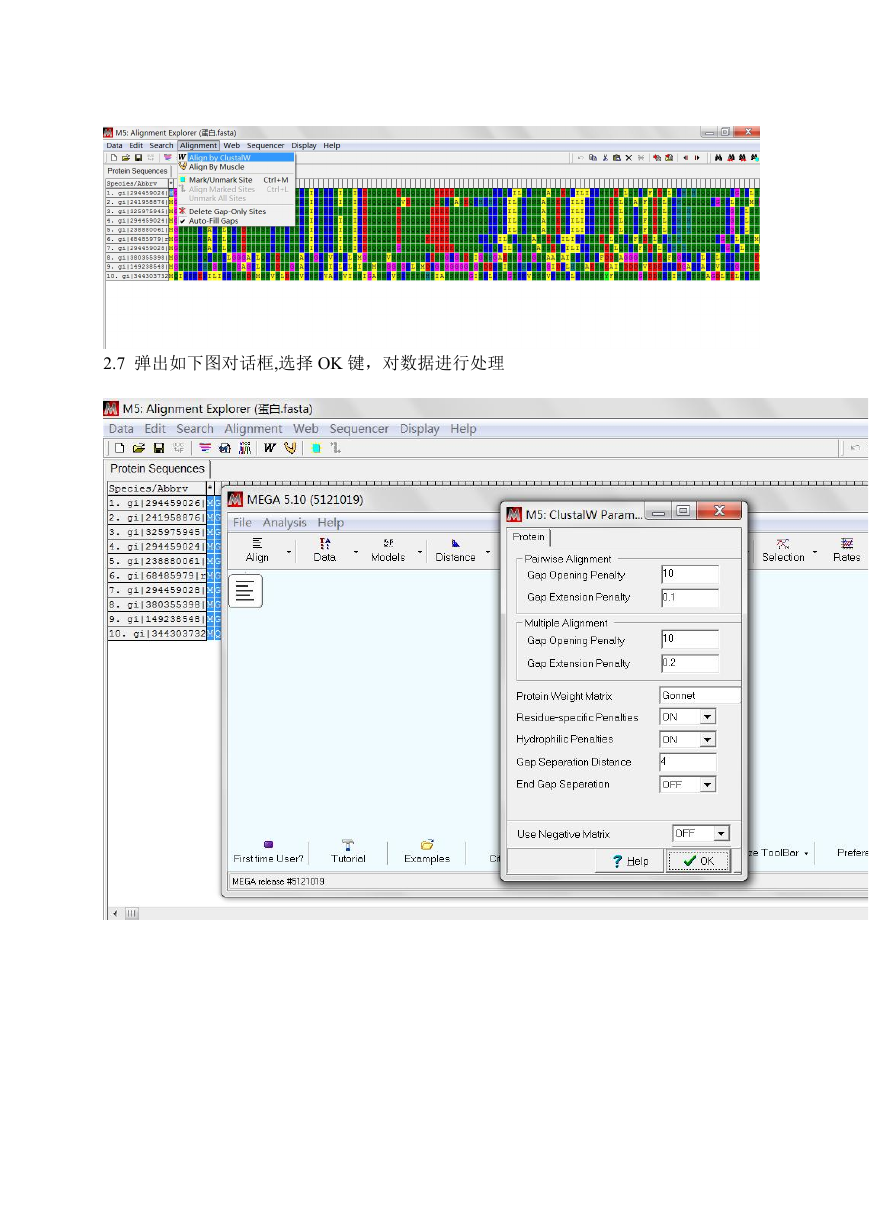
2.6 点击菜单栏内的 Alignment 选项,选择 Align by ClustalW 选项。
�
2.7 弹出如下图对话框,选择 OK 键,对数据进行处理
�
经过一段时间的数据处理,数据处理完成如下图所示:
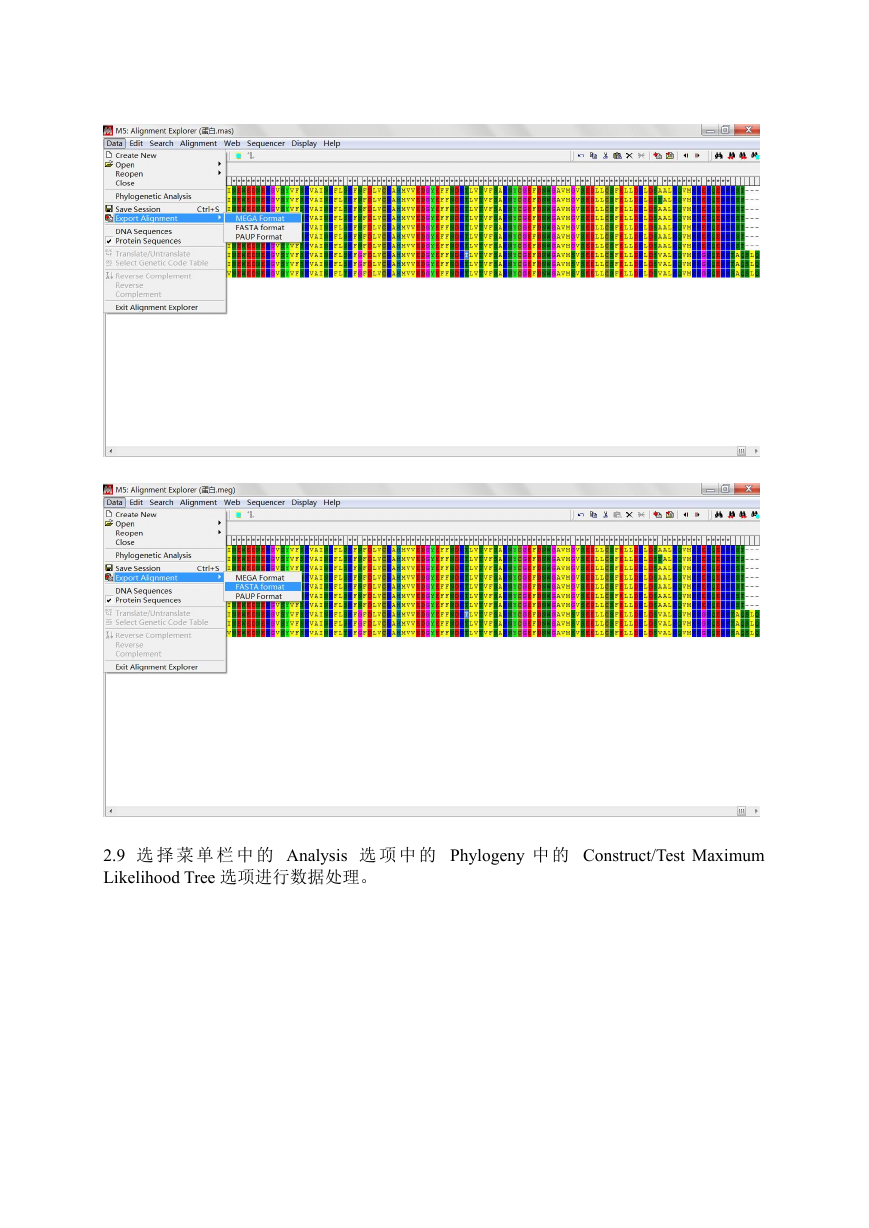
2.8 选择菜单栏中 Data 选项中的 Save Session 选项进行保存。
再选择 Export Alignment 中的 MEGA Format 和 FASTA format 进行保存。
�
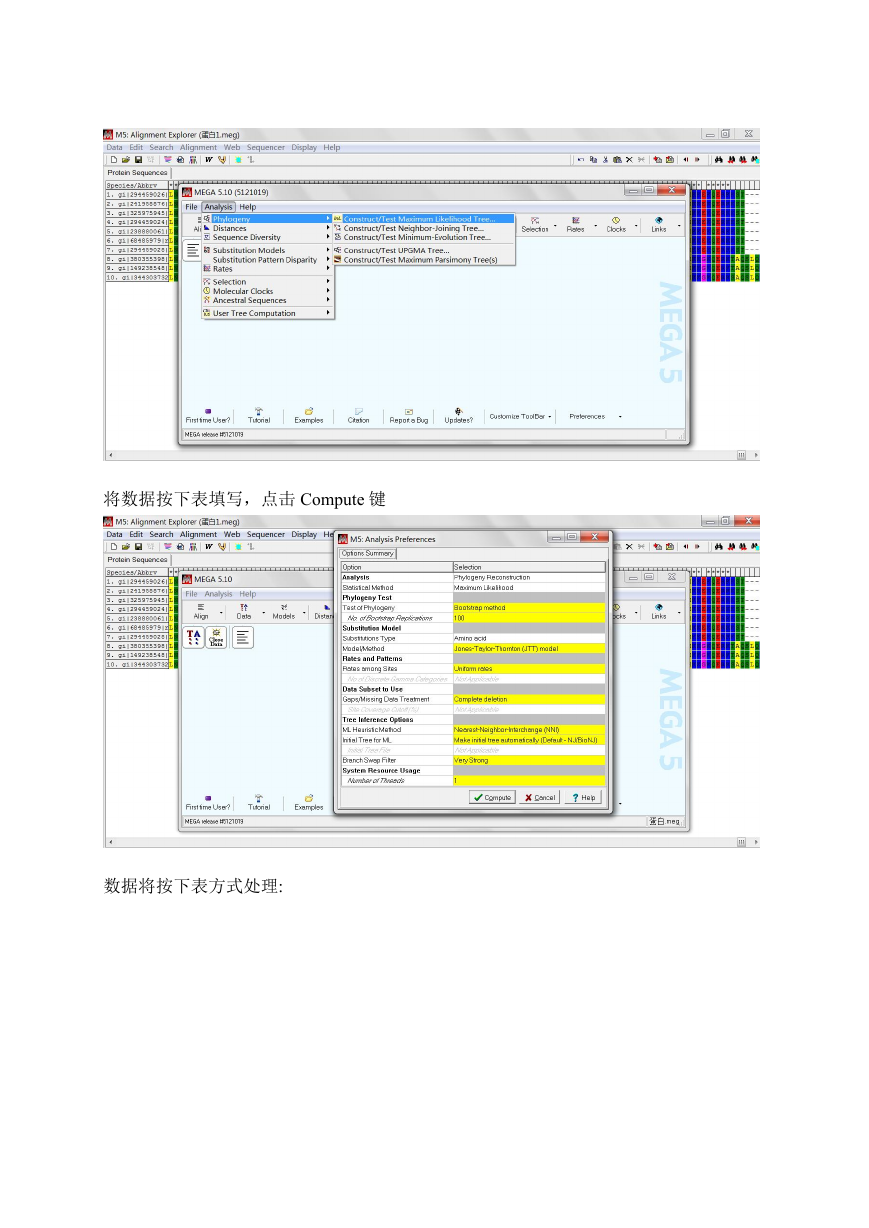
2.9 选 择 菜 单 栏 中 的 Analysis 选 项 中 的 Phylogeny 中 的 Construct/Test Maximum
Likelihood Tree 选项进行数据处理。
�
将数据按下表填写,点击 Compute 键
数据将按下表方式处理:
�











 2023年江西萍乡中考道德与法治真题及答案.doc
2023年江西萍乡中考道德与法治真题及答案.doc 2012年重庆南川中考生物真题及答案.doc
2012年重庆南川中考生物真题及答案.doc 2013年江西师范大学地理学综合及文艺理论基础考研真题.doc
2013年江西师范大学地理学综合及文艺理论基础考研真题.doc 2020年四川甘孜小升初语文真题及答案I卷.doc
2020年四川甘孜小升初语文真题及答案I卷.doc 2020年注册岩土工程师专业基础考试真题及答案.doc
2020年注册岩土工程师专业基础考试真题及答案.doc 2023-2024学年福建省厦门市九年级上学期数学月考试题及答案.doc
2023-2024学年福建省厦门市九年级上学期数学月考试题及答案.doc 2021-2022学年辽宁省沈阳市大东区九年级上学期语文期末试题及答案.doc
2021-2022学年辽宁省沈阳市大东区九年级上学期语文期末试题及答案.doc 2022-2023学年北京东城区初三第一学期物理期末试卷及答案.doc
2022-2023学年北京东城区初三第一学期物理期末试卷及答案.doc 2018上半年江西教师资格初中地理学科知识与教学能力真题及答案.doc
2018上半年江西教师资格初中地理学科知识与教学能力真题及答案.doc 2012年河北国家公务员申论考试真题及答案-省级.doc
2012年河北国家公务员申论考试真题及答案-省级.doc 2020-2021学年江苏省扬州市江都区邵樊片九年级上学期数学第一次质量检测试题及答案.doc
2020-2021学年江苏省扬州市江都区邵樊片九年级上学期数学第一次质量检测试题及答案.doc 2022下半年黑龙江教师资格证中学综合素质真题及答案.doc
2022下半年黑龙江教师资格证中学综合素质真题及答案.doc