Discovery Studio3.0 分子动力学教程
Discovery Studio Molecular Dynamics 教程
Molecular Dynamics – 分子动力学方法
目的:采用 Discovery Studio,以一组蛋白-配体复合物为实例,示范分子动力学及结果分析
操作过程。
所需功能和模块:Discovery Studio Visualizer client, CHARMm, Simulation protocols
所需数据文件:1IE9_VDX.dsv
所需时间:1 小时
介绍
分子动力学(Molecular Dynamics, MD)是分子模拟中最常用的方法之一。该方法基于分
子力场,能够动态的描述分子的运动状况,继而描述生命的动态过程。分子动力学在生命科
学领域中的应用非常广泛,如蛋白质折叠的机理研究、酶催化反应的机理研究、与功能相关
蛋白质的运动研究、生物大分子大范围构象变化的研究等等。近几十年来,分子动力学方法
已经成功的运用于大分子体系低能量构象的模建、X 射线晶体衍射以及 NMR 实验结果的处
理。[1,2] 现在分子动力学方法已经成了理论生物学研究中必不可少的方法之一。[3,4]
分子动力学模拟主要包括如下几个步骤:
1. 模拟体系升温过程(Heating Stage)
2. 模拟体系平衡过程(Equilibrium Stage)
3. 模拟体系采样过程(Production Stage)
4. 分析体系的目标性质(Analysis)
然而,由于一般的待模拟体系的初始结构或多或少的存在缺陷(比如,初始结构不完整
或存在不合理的结构区域),所以我们往往需要对初始结构进行预处理才能进行分子动力学
模拟。一个完整的分子动力学模拟过程应包括如下几个步骤:
1. 初始结构检查及预处理
2. 给模拟体系赋力场参数
3. 考虑溶剂效应
4. 初始结构能量最小化
5. 动力学模拟(包括升温、平衡、采样)
6. 结果分析
目前,常用的分子动力学模拟软件都基于 Unix(Linux)操作系统。实施每一个模拟的
步骤都需要特定的命令来调用相关的程序对模拟体系进行处理。同时,模拟者还需要熟知每
个参数的意义并定义相关的参数值。最终分析过程也需要配合其他软件才能完成。这对初级
分子动力学模拟者而言是非常困难的(模拟者不仅需要掌握分子动力学软件的使用命令,还
需要掌握操作系统相关的命令)。为了解决这样的问题,Discovery Studio 为用户提供了基于
窗口的分子动力学模拟工具。使用 DS,用户可以轻松的完成分子动力学的整个模拟过程(包
括体系的准备、模拟过程的设置及结果的分析)。另外考虑到初级模拟者在整个模拟流程方
面不熟悉,DS 专门设置了一个“Standard Dynamics Cascade”protocol。该 Protocol 将整个
动力学模拟的步骤集合到一个参数设置界面当中,方便初级模拟者的使用。
为了使大家对 MD 的操作流程有所了解,本教程主要内容包括如下几个方面:
1. 初始结构的准备,力场设置
1
�
Discovery Studio3.0 分子动力学教程
2. 对结构进行溶剂化处理
3. 标准的分子动力学模拟流程
4. 动力学模拟结果的轨迹分析
以下以一组蛋白-配体复合物为例进行部分束缚的动力学模拟,介绍 Discovery Studio 中
分子动力学的基本操作方法:
准备蛋白-配体复合物
选择并定义原子组
添加溶剂
设置束缚部分
进行带束缚的分子动力学模拟
分析动力学模拟轨迹
计算蛋白-配体相互作用能量
准备分子动力学体系,执行分子动力学计算
1. 导入蛋白-配体复合物,并赋予力场参数
双击图标,启动 Discovery Studio,在文件浏览器(Files Explorer)找到 1IE9_VDX.dsv 文件,
双击打开,将蛋白-配体复合物导入 3D 窗口。(图 1)
展开 Simulation 工具栏,在工具浏览器(Tools Explorer)中展示分子动力学模拟工具组。
展开 Change Forcefield 工具面板,缺省力场为 CHARMm 力场。若需其他力场,可通过
Forcefield 下拉菜单选择,然后点击 Apply Forcefield 命令,给蛋白-配体复合物赋力场。清
除当前力场点击 Clear Forcefield 命令。(图 1)
图 1 复合物的三维结构及赋予复合物力场参数
2. 添加溶剂环境
为使动力学模拟过程更接近真实情况,可模拟人体内环境添加溶剂环境。
从 Protocol 面板组中找到 Simulation | Dyamics 文件夹,点击 Solvation 流程(图 2),该流程
在参数浏览器(Parameters Explorer)中打开。
2
�
Discovery Studio3.0 分子动力学教程
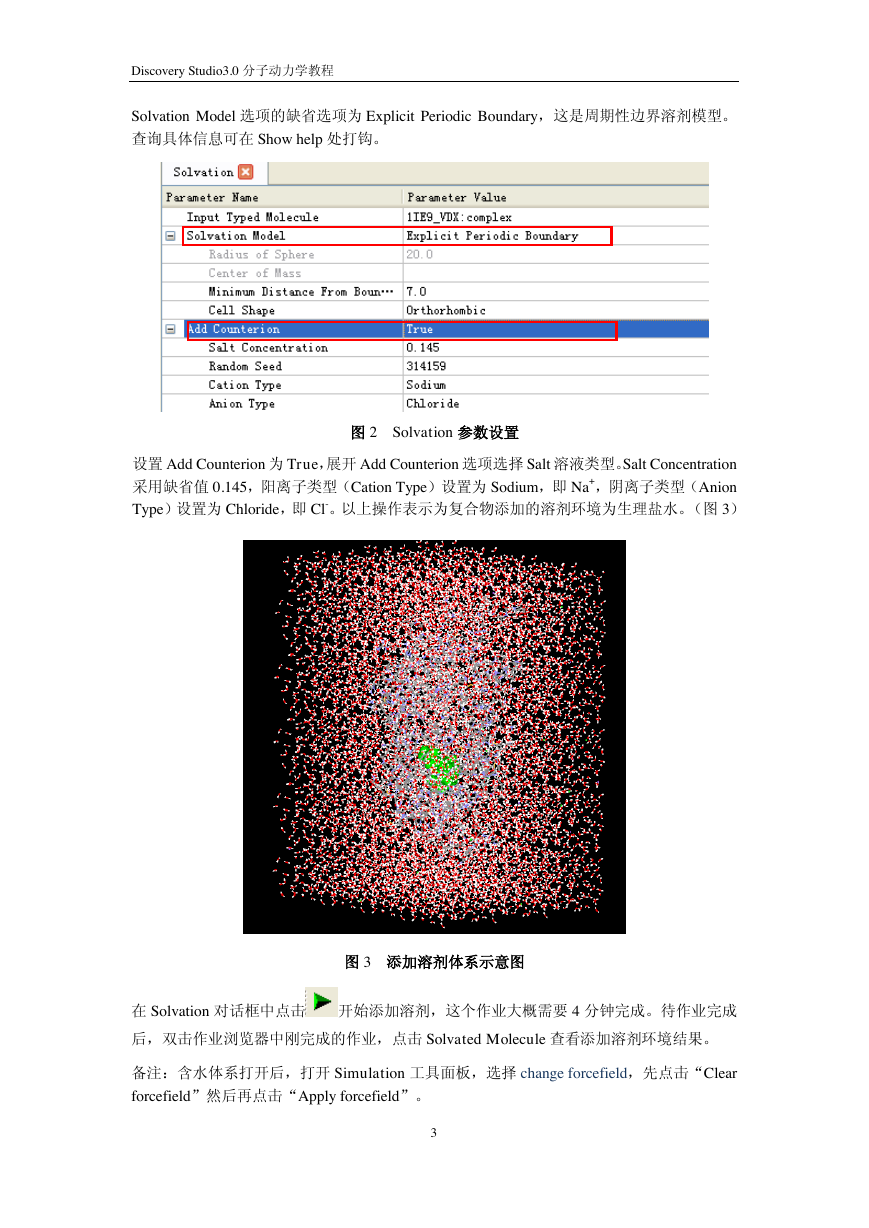
Solvation Model 选项的缺省选项为 Explicit Periodic Boundary,这是周期性边界溶剂模型。
查询具体信息可在 Show help 处打钩。
图 2 Solvation 参数设置
设置 Add Counterion 为 True,展开 Add Counterion 选项选择 Salt 溶液类型。Salt Concentration
采用缺省值 0.145,阳离子类型(Cation Type)设置为 Sodium,即 Na+,阴离子类型(Anion
Type)设置为 Chloride,即 Cl-。以上操作表示为复合物添加的溶剂环境为生理盐水。(图 3)
图 3 添加溶剂体系示意图
在 Solvation 对话框中点击
开始添加溶剂,这个作业大概需要 4 分钟完成。待作业完成
后,双击作业浏览器中刚完成的作业,点击 Solvated Molecule 查看添加溶剂环境结果。
备注:含水体系打开后,打开 Simulation 工具面板,选择 change forcefield,先点击“Clear
forcefield”然后再点击“Apply forcefield”。
3
�
Discovery Studio3.0 分子动力学教程
3. 设置束缚
由于添加溶剂时,水分子和离子都是规则排列,此教程中,我们在进行动力学时首先束缚住
蛋白-配体复合物体系,仅优化一下水分子和离子,所以需要设置束缚体系。在系统升温和
平衡的时候,我们希望蛋白-配体复合物能有一定的自由度,于是我们选择逐渐减小对系统
的束缚,这一点可以通过 DS 中的 Harmonic Restraint 来实现。
系统视图中点击选中受体分子 Receptor,按住 Ctrl 键同时选择配体分子 VDX,下拉菜单中
选择 Edit | Group„命令,在 Group 对话框中输入 Group 的名字“Fixed”,点击 Defined 定义
名字。点击 3D 窗口任意空白处取消选择。新定义的组将出现在系统视图中。以同样的方式
我们定义两个 Constraint 组,分别为 Constraint1 和 Constraint2。
选中 Fixed,在 Simulation 工具面板里 Setup Constraints 中,点击“Create Fixed Atom
Constraint”。选中 Constraint1,在 Simulation 工具面板里 Setup Constraints 中,点击“Create
Harmonic Restraint”,并在 Table 中将“Force Constant”由 10 改为 50。选中 Constraint2,在
Simulation 工具面板里 Setup Constraints 中,点击“Create Harmonic Restraint”。
约束结果显示如图 5.
图 4 设置束缚体系
点击 3D 窗口任意空白处取消选择。3D 窗口中可以看到被束缚的原子周围环绕着蓝色的小
槛(图 5)。
4
�
Discovery Studio3.0 分子动力学教程
图 5 设好束缚的体系
5. 打开动力学流程,修改参数
确保添加了溶剂体系的 complex.dsv 窗口为当前活跃窗口,文件流程浏览器(Protocol
Explorer)中双击 Simulations 文件夹下 Standard Dynamics Cascade 流程,该流程在参数浏览
器(Parameters Explorer)中打开,“complex:complex”自动载入到 Input Typed Molecule 参
数格。
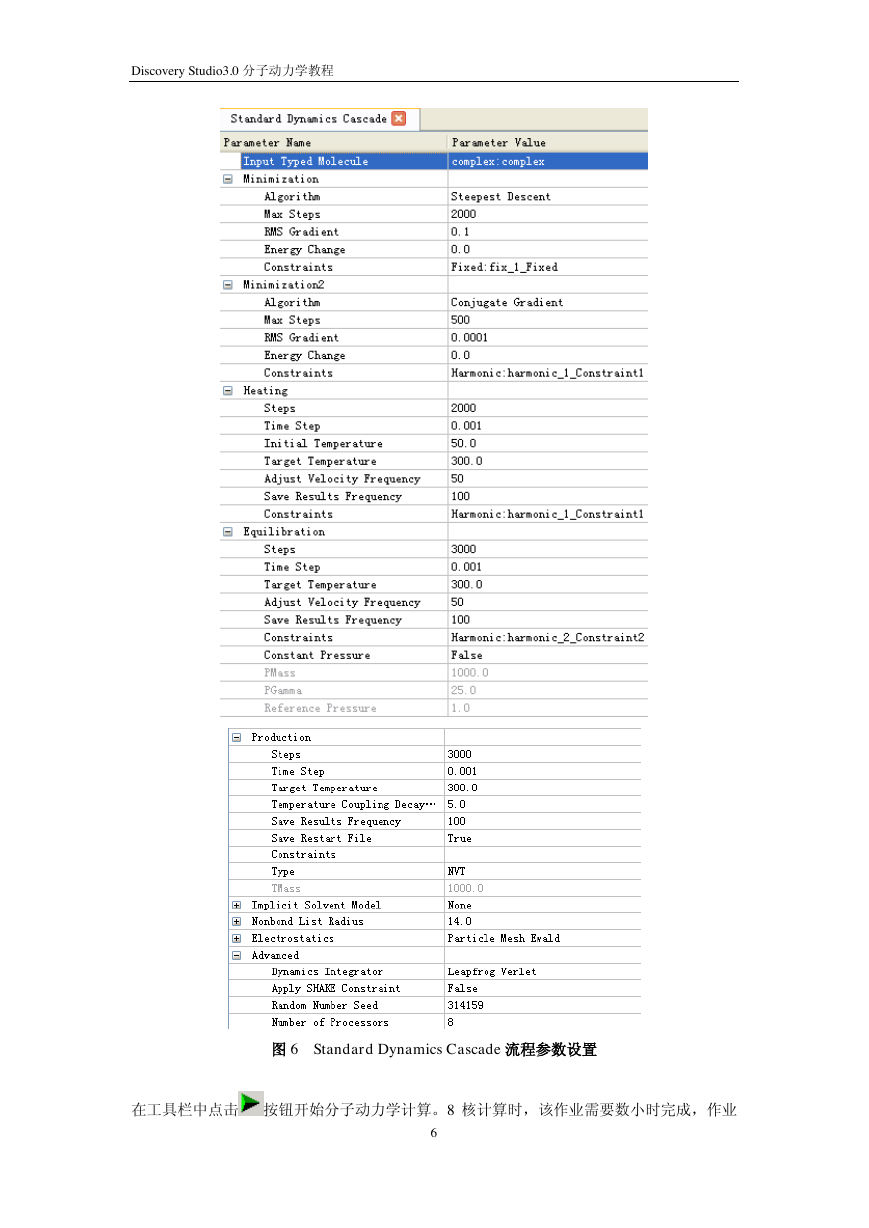
Standard Dynamics Cascade 流程包括 5 个阶段:优化 I(Minimization)、优化 II(Minimization2)、
升温(Heating)、平衡(Equilibration)、模拟采样(Production)。
展开 Minimization 阶段的参数组,确保 Constraints 选项选择“Fixed:fix_1_Fixed”。
展 开 Minimization 阶 段 的 参 数 组 , 确 保 Constraints 选 项 选 择
“Harmonic:harmonic_1_Constraint1”。
展 开 Heating 阶 段 的 参 数 组 , 确 保 Constraints 选 项 选 择
“Harmonic:harmonic_1_Constraint1”。
展 开 Equilibration 阶 段 的 参 数 组 , 确 保 Constraints 选 项 选 择
“Harmonic:harmonic_1_Constraint1”。并确保 Constrant Pressure 为 True。
展开 Prodution 阶段参数组,设置 Steps 为 5000,步长 Time Step 为 0.002,Save Results Frequency
为 100,Type 为 NPT。设置 Implicit Solvent Modeler 选项设置为 None,Electrostatics 选项选
择 Particle Mesh Ewald。Apply Shake Constraint 为 True。Save Restart File 为 True(图
5
�
Discovery Studio3.0 分子动力学教程
图 6 Standard Dynamics Cascade 流程参数设置
在工具栏中点击 按钮开始分子动力学计算。8 核计算时,该作业需要数小时完成,作业
6
�
Discovery Studio3.0 分子动力学教程
的状态可以在“Jobs Explorer”中查看。当任务完成对话框打开,点击 OK 关闭。
6. 查看结果,显示构象变化动画
备注:对于长时间的分子动力学,需要大量的内存将所产生的所有动力学轨迹文件加入到输
出的.dsv 格式的文件中。如果 server 的内存不够,.dsv 文件将无法及时将.dcd 格式的动力学
轨迹文件中加入。对于这种情况,请下文介绍的方法载入动力学轨迹文件,显示蛋白体系构
象变化的动画。
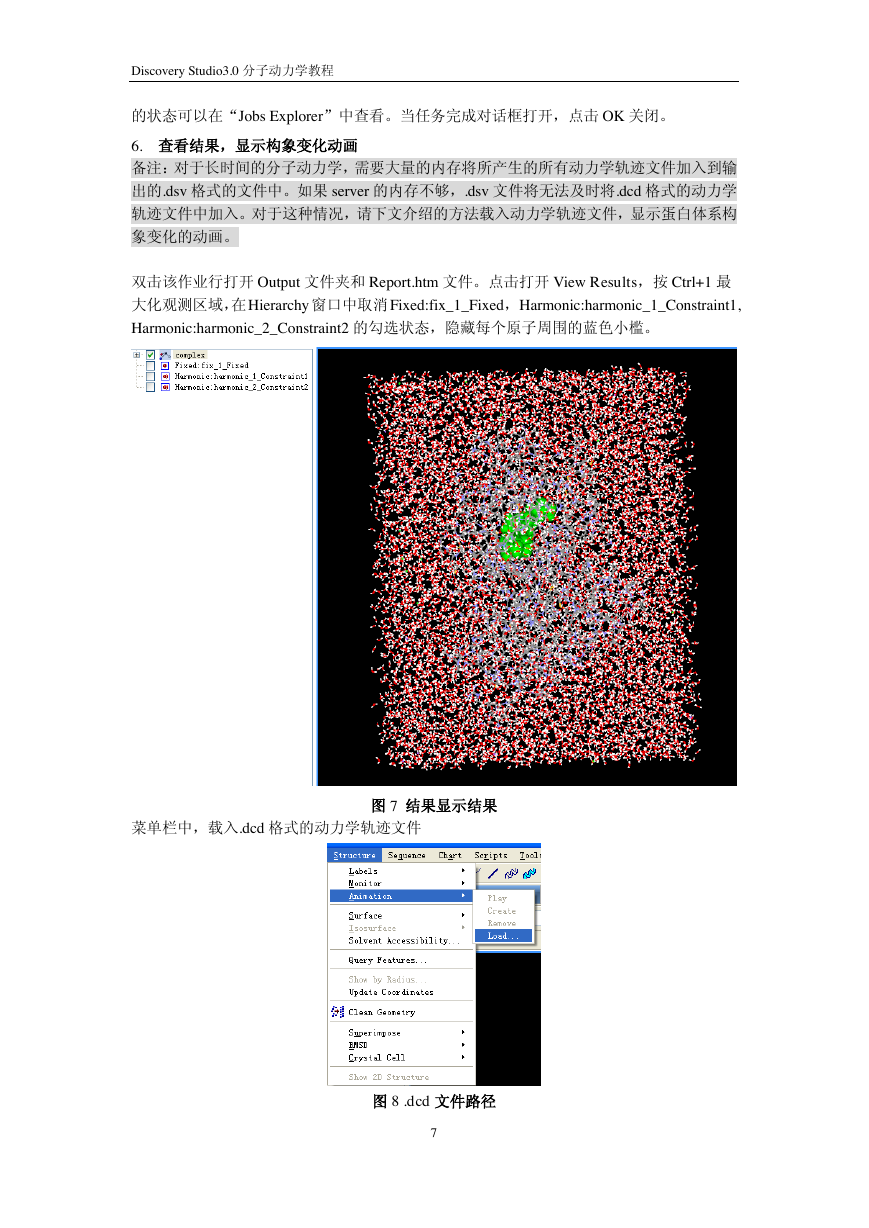
双击该作业行打开 Output 文件夹和 Report.htm 文件。点击打开 View Results,按 Ctrl+1 最
大化观测区域,在 Hierarchy 窗口中取消 Fixed:fix_1_Fixed, Harmonic:harmonic_1_Constraint1,
Harmonic:harmonic_2_Constraint2 的勾选状态,隐藏每个原子周围的蓝色小槛。
菜单栏中,载入.dcd 格式的动力学轨迹文件
图 7 结果显示结果
图 8 .dcd 文件路径
7
�
Discovery Studio3.0 分子动力学教程
确保 3D 窗口是当前活跃窗口,展开 Hierarchy 窗口中的 complex, 分别选择 W(water),CAL
(Cl-), SOD(Na+),在右键菜单中选择 Hide。选择下拉菜单 Structure | Animation | Play
命令,则可以看到在 3D 窗口中的复合物开始运动,显示动力学模拟过程中的变化。动画循
环往复播放。再次选择下拉菜单 Structure | Animation | Play 命令可以停止动画播放。
分析分子动力学计算结果
1. 绘制能量曲线
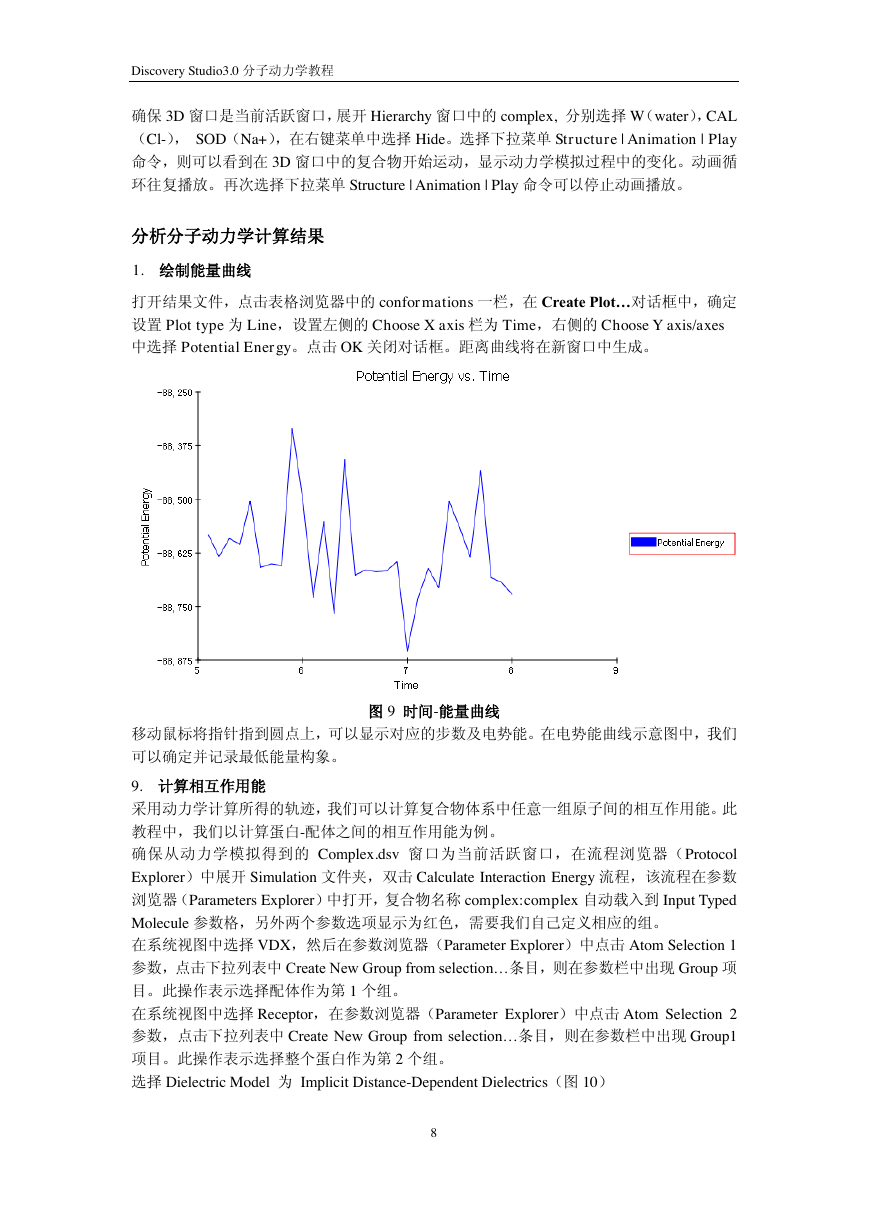
打开结果文件,点击表格浏览器中的 conformations 一栏,在 Create Plot…对话框中,确定
设置 Plot type 为 Line,设置左侧的 Choose X axis 栏为 Time,右侧的 Choose Y axis/axes
中选择 Potential Energy。点击 OK 关闭对话框。距离曲线将在新窗口中生成。
图 9 时间-能量曲线
移动鼠标将指针指到圆点上,可以显示对应的步数及电势能。在电势能曲线示意图中,我们
可以确定并记录最低能量构象。
9. 计算相互作用能
采用动力学计算所得的轨迹,我们可以计算复合物体系中任意一组原子间的相互作用能。此
教程中,我们以计算蛋白-配体之间的相互作用能为例。
确保从动力学模拟得到的 Complex.dsv 窗口为当前活跃窗口,在流程浏览器(Protocol
Explorer)中展开 Simulation 文件夹,双击 Calculate Interaction Energy 流程,该流程在参数
浏览器(Parameters Explorer)中打开,复合物名称 complex:complex 自动载入到 Input Typed
Molecule 参数格,另外两个参数选项显示为红色,需要我们自己定义相应的组。
在系统视图中选择 VDX,然后在参数浏览器(Parameter Explorer)中点击 Atom Selection 1
参数,点击下拉列表中 Create New Group from selection…条目,则在参数栏中出现 Group 项
目。此操作表示选择配体作为第 1 个组。
在系统视图中选择 Receptor,在参数浏览器(Parameter Explorer)中点击 Atom Selection 2
参数,点击下拉列表中 Create New Group from selection…条目,则在参数栏中出现 Group1
项目。此操作表示选择整个蛋白作为第 2 个组。
选择 Dielectric Model 为 Implicit Distance-Dependent Dielectrics(图 10)
8
�












 2023年江西萍乡中考道德与法治真题及答案.doc
2023年江西萍乡中考道德与法治真题及答案.doc 2012年重庆南川中考生物真题及答案.doc
2012年重庆南川中考生物真题及答案.doc 2013年江西师范大学地理学综合及文艺理论基础考研真题.doc
2013年江西师范大学地理学综合及文艺理论基础考研真题.doc 2020年四川甘孜小升初语文真题及答案I卷.doc
2020年四川甘孜小升初语文真题及答案I卷.doc 2020年注册岩土工程师专业基础考试真题及答案.doc
2020年注册岩土工程师专业基础考试真题及答案.doc 2023-2024学年福建省厦门市九年级上学期数学月考试题及答案.doc
2023-2024学年福建省厦门市九年级上学期数学月考试题及答案.doc 2021-2022学年辽宁省沈阳市大东区九年级上学期语文期末试题及答案.doc
2021-2022学年辽宁省沈阳市大东区九年级上学期语文期末试题及答案.doc 2022-2023学年北京东城区初三第一学期物理期末试卷及答案.doc
2022-2023学年北京东城区初三第一学期物理期末试卷及答案.doc 2018上半年江西教师资格初中地理学科知识与教学能力真题及答案.doc
2018上半年江西教师资格初中地理学科知识与教学能力真题及答案.doc 2012年河北国家公务员申论考试真题及答案-省级.doc
2012年河北国家公务员申论考试真题及答案-省级.doc 2020-2021学年江苏省扬州市江都区邵樊片九年级上学期数学第一次质量检测试题及答案.doc
2020-2021学年江苏省扬州市江都区邵樊片九年级上学期数学第一次质量检测试题及答案.doc 2022下半年黑龙江教师资格证中学综合素质真题及答案.doc
2022下半年黑龙江教师资格证中学综合素质真题及答案.doc